- タンパク質コード遺伝子にみられる3種類の転写後制御を説明せよ
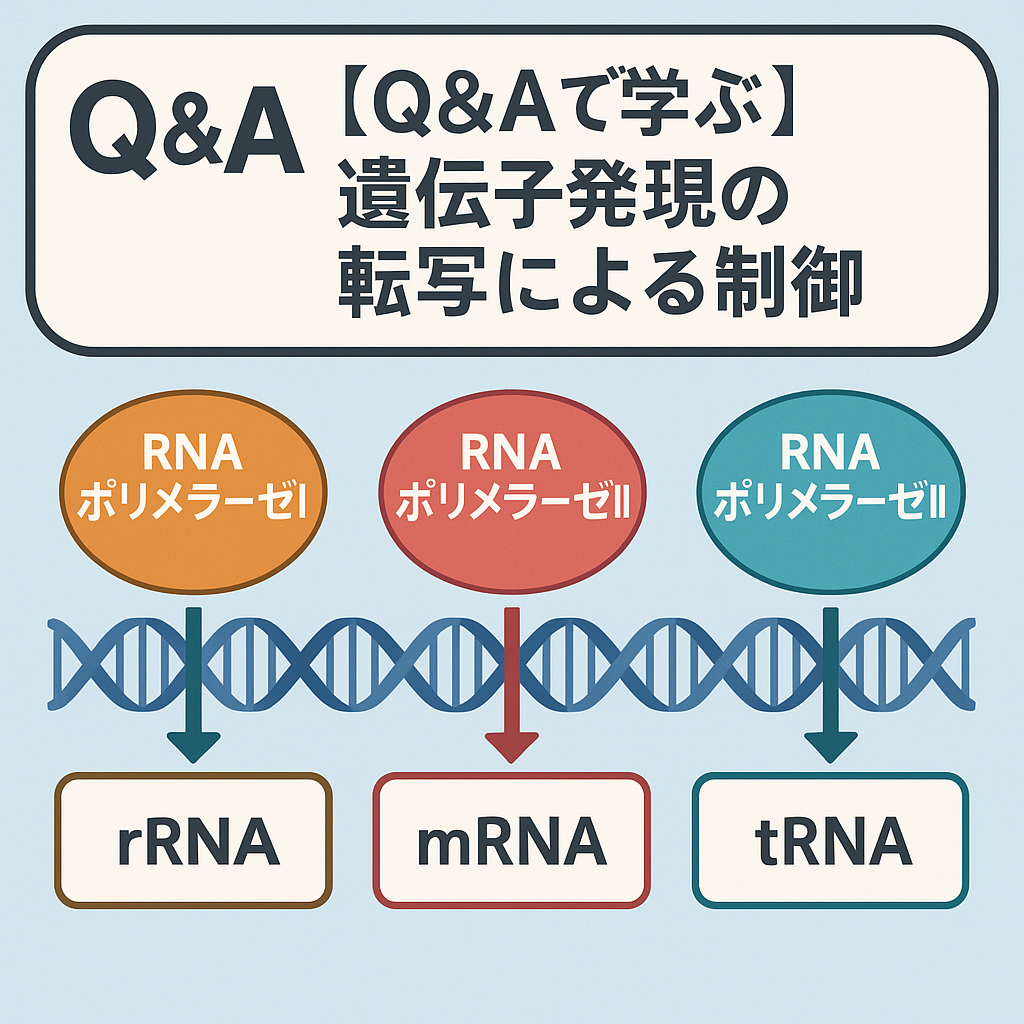
- 今,最近発見された真核生物の遺伝子の転写制御機構を調べているとしよう。この生物には3種類のRNA ポリメラーゼ様の酵素があり、タンパク質をコードする遺伝子は RNAポリメラーゼⅡ様の酵素によって転写されることを見いだした。生成した完全長のmRNAを調べたところ、それには5’構造(つまり、キャップ)がないことがわかった。そのことに驚くだろうか。なぜ驚くか、あるいはなぜ驚かないか。
- RNAポリメラーゼIIによる転写終結とポリアデニル酸付加が共役している証拠は何か
- HIVの抗終結機構を操作することによって,AIDSとの闘いに効果的な治療法をもたらす可能性が示唆されている。Tat タンパク質の結合を失わせるTAR 配列の変異は、HIV感染後の HIVの転写にどのような影響を与えるか、またそれはなぜか。活性を失わせるCdk9の変異はどうか
- スプライシングの際にイントロンが除去されることは、どのようにして発見されたか。エキソンーイントロン連結部の位置は、どうすれば予測できるか
- hnRNA, snRNA, miRNA, siRNA, snoRNAの違いは何か
- グループⅡイントロンの自己スプライシングと、スプライソソームのスプライシングとの機構上の類似点は何か。この二つの間に進化上の関係を示す証拠は何か
- 研究者は、転写とRNAプロセシングはどこで起こると信じているか。これを支持する証拠は何か
- 10個のエキソン、9個のイントロン、およびポリアデニル酸付加共通配列をもつ3’UTRを含む遺伝子の配列が決定されたとしょう。5番目のイントロンにもポリアデニル酸付加部位が存在した。二つのポリアデニル酸付加部位がともに使われるかどうかを調べるため、mRNAを単離したところ、筋肉組織からは長い転写産物が、他のすべての組織からは短いmRNA転写産物が見いだされたとする。これら二つの転写産物の生成に関与する機構を推測せよ。
- RNA編集は、トリパノソーマや植物のミトコンドリア、葉緑体、そして、まれには高等真核生物で起こる基本的な現象である。RNA編集とは何か。また,ヒトのアポBの例で明らかになっている、この現象がもたらす利点は何か。
- 核内のタンパク質複合体は、mRNA分子を細胞質へ輸送する役割をもっている。核外輸送因子複合体を形成するタンパク質について説明せよ。また、mRNPと複合体が細胞質への方向性で動く機構の背景にある二群のタンパク質は何か。
- RNAノックダウンは、遺伝子発現を変える一連の方法のなかでも有力な道具である。どのように遺伝子発現がノックダウンされるかを簡単に述べよ.TSC1に対する siRNAをヒト細胞に導入したときの効果を述べよ。
- Dicer活性を欠く植物では、なぜRNAウイルスの感染に対する感受性が高まるのかを推測せよ
- ある種のmRNAが,細胞内の特異的な場所に蓄積するようになっていることを示す証拠は何か
タンパク質コード遺伝子にみられる3種類の転写後制御を説明せよ
タンパク質をコードする遺伝子の発現量や機能は、転写後(mRNAが合成された後)の段階で多段階に制御されています。代表的な「3種類の転写後制御」は次の通りです。
1. mRNAプロセシング(選択的スプライシング)
- 説明
合成された一次転写産物(pre-mRNA)は、エキソンとイントロンから構成されている。イントロン配列はスプライシングにより除去され、エキソンのみが連結されて成熟mRNAとなる。 - 選択的スプライシングと呼ばれる現象では、特定のエキソンが選択的に残されたり除かれたりすることで、1つの遺伝子から複数種のmRNAおよび、それに対応した異なるタンパク質多様体(アイソフォーム)が生み出される。
- この機構により、生物は遺伝子数に比して格段に多様なタンパク質を産生できる。
2. mRNAの安定性・分解制御
- 説明
細胞内のmRNAは安定性が制御されており、mRNAの寿命に応じて翻訳されるタンパク質の量が大きく影響される。mRNAの3’末端(ポリアデニル化部位)や5’末端(キャップ構造)、一部の配列(AUリッチエレメントなど)に結合するタンパク質やmiRNAが、分解酵素を呼び寄せたり、安定化したりして調節している。 - たとえば、特定のシグナルが作用したときに迅速かつ大量のタンパク質合成を行いたい場合、安定なmRNAが利用される。一方、非常に短命なmRNAを用いることで、一過性の発現も実現可能になる。
3. 翻訳調節(トランスレーショナルコントロール)
- 説明
mRNAが細胞質に運ばれた後、リボソームによってタンパク質へと翻訳される。この段階でも5’UTRや3’UTRの特定配列、miRNA・RNA結合タンパク質による結合、または開始因子の調整などによって、mRNAの翻訳効率が制御される。 - たとえば、ストレス応答時には全体的な翻訳抑制因子(eIF2αのリン酸化など)が働いて特定のmRNA以外の翻訳が抑えられたり、逆に特定mRNAだけが選択的に翻訳されたりする。
補足
これらの転写後制御は単独で働く場合もあれば、複数が組み合わさって遺伝子発現調節の精密な制御を実現しています。したがって「選択的スプライシング」「mRNA安定性・分解制御」「翻訳調整」の3つは、タンパク質コード遺伝子の発現調節における柱となっています。
今,最近発見された真核生物の遺伝子の転写制御機構を調べているとしよう。この生物には3種類のRNA ポリメラーゼ様の酵素があり、タンパク質をコードする遺伝子は RNAポリメラーゼⅡ様の酵素によって転写されることを見いだした。生成した完全長のmRNAを調べたところ、それには5’構造(つまり、キャップ)がないことがわかった。そのことに驚くだろうか。なぜ驚くか、あるいはなぜ驚かないか。
一般の真核生物において、タンパク質をコードするmRNAはRNAポリメラーゼIIによって転写され、そのmRNAの5’末端には「7-メチルグアノシンキャップ(5’キャップ)」が必ず付加されます。これは転写開始直後にキャッピング酵素複合体によって化学的に修飾される特徴的な構造であり、mRNAの安定性や核外輸送、翻訳開始の効率・正確さに極めて重要な役割を果たします。
なぜ「驚く」と考えられるか
- 通常ならありえない現象
- 真核生物で「タンパク質コード遺伝子のmRNAに5’キャップが無い」という例は、原則として報告がありません。「5’キャップ構造」は真核生物のmRNAの普遍的特徴だからです。
- 5’キャップはリボソームによる翻訳開始に必須となるため、これが無い場合は通常、mRNAは翻訳に使われにくく、また分解されやすくなってしまいます。
- RNAポリメラーゼII(またはII様酵素)とのカップリング
- mRNAのキャッピングは「RNAポリメラーゼIIのCTD(C末端反復ドメイン)」を足場としてキャッピング酵素が結合することによって起こるため、ポリメラーゼⅡ様酵素にその足場や機能が無い場合、キャップが付加されない可能性があります。
- これがすなわち「ポリメラーゼの種類によってはキャッピング機構自体が進化的に欠落している」可能性を示唆します。
なぜ「驚かない」と考える場合
- 特殊な生物や例外的なメカニズムが存在する可能性
- 極めて特殊な進化をした真核生物や、既知とは異なる転写・翻訳装置を持つ生物であれば、「5’キャップが無いmRNAでも安定/翻訳可能」な例外が進化した可能性も、完全には否定できません。
まとめ
- 本来なら「非常に驚く」現象です。
- 真核生物のタンパク質コードmRNAは5’キャップを持つのが基本であり、その機能的意義も大きいからです。
- もしmRNAに5’キャップが観察されない場合は、「この酵素(RNAポリメラーゼロ様酵素)や生物系で未知のmRNA修飾や翻訳開始機構がある」「キャッピングの役割を担う別の機構が進化している」など、教科書的理解を覆す現象であるため、注目に値します。
- まれな例外や特殊環境型生物であれば「驚かない」という立場もあり得ますが、現時点での生物学の常識からは「非常に驚く」と結論できます。
RNAポリメラーゼIIによる転写終結とポリアデニル酸付加が共役している証拠は何か
RNAポリメラーゼIIによる転写終結(トランスクリプション・ターミネーション)とmRNAの3’末端でのポリアデニル酸(polyA)付加は、単なる連続的な反応ではなく、分子レベルで密接に結びついていることが多くの実験から明らかになっています。主な証拠には以下のようなものがあります。
1. ポリアデニル化(polyA)シグナルの機能が転写終結に不可欠
- 多くの研究で、ポリアデニル化シグナル(AAUAAA配列など)を欠損させると、適切な転写終結が起こらず、RNAポリメラーゼIIが通常よりもはるか下流まで転写を続けてしまうことが示されています。
- 逆に、正常なポリアデニル化シグナルが存在すると、その直後で転写が効率よく終結します。
2. 終結と3’末端形成(ポリアデニル酸付加)のための因子が物理的に結合
- ポリA付加や切断に関わる因子(CPSF、CstFなど)は、RNAポリメラーゼIIのC末端ドメイン(CTD)と物理的に結合し、転写伸長中の複合体内で両反応を協調して制御しています。
- これにより、「3’切断・ポリアデニル酸付加」と「ポリメラーゼの離脱=転写終結」が分子的に同時進行しやすくなります。
3. ポリAシグナルでmRNAが切断されると、下流のRNA断片が分解されてポリメラーゼIIの解離が誘導される(トーピードモデル)
- mRNAがポリアデニル化シグナルで切断されると、下流側(ポリメラーゼに続く側)のRNA断片は5’→3’エキソヌクレアーゼ(例:Xrn2)によって分解されます。このエキソヌクレアーゼがRNAポリメラーゼIIに「追いつく」と、ポリメラーゼはDNAから解離します。
- この現象は「トーピードモデル」と呼ばれ、3’末端形成(=ポリアデニル化)と転写終結が直接的に連動していることを示します。
4. in vitro系や遺伝学的変異体の解析
- in vitroでの再構成実験でも、転写中のRNAポリメラーゼIIがポリAシグナルで一時停止し、その後mRNAが切断・ポリアデニル化された時点でポリメラーゼがDNAから離脱することが証明されています。
- 3’末端の形成因子に温度感受性やノックアウト・ミュータントを作ると、転写終結も同時に障害されるため、両者の共役が必須であることが分かります。
まとめ
RNAポリメラーゼIIによる転写終結とポリアデニル酸付加は、(1)ポリAシグナルの有無による終結のコントロール、(2)付加因子の複合体形成、(3)下流断片分解によるポリメラーゼの離脱、(4)変異やin vitro実験による共役性の証明などから、オーケストラのように密接に連動していることが明らかになっています。
こうした分子機構は「生体内でmRNA 3’末端形成と転写終結が同時進行する」ための重要な進化的仕組みです。
HIVの抗終結機構を操作することによって,AIDSとの闘いに効果的な治療法をもたらす可能性が示唆されている。Tat タンパク質の結合を失わせるTAR 配列の変異は、HIV感染後の HIVの転写にどのような影響を与えるか、またそれはなぜか。活性を失わせるCdk9の変異はどうか
TAR配列の変異(Tatタンパク質の結合喪失)がHIV転写に及ぼす影響と理由
HIVでは、Tatタンパク質が「TAR配列」と呼ばれるRNA構造に特異的に結合し、転写伸長過程を促進します。TAR配列に変異が入り、Tatタンパク質が結合できなくなると、以下のような影響が起きます。
- HIVの転写効率が大幅に低下し、ウイルス遺伝子の発現が著しく減少します。
これは、TatがTAR配列を介して転写複合体に結合することが、転写開始後まもなく“停止”しているRNAポリメラーゼIIを再活性化し、長いmRNAを合成させるために必須だからです。 - TARが変異した場合、「Pause」状態のポリメラーゼIIが復帰できず、ごく短い転写産物しかできなくなり、HIV遺伝子は十分に発現されません。
理由:
- TatはTARと結合したうえでP-TEFb(Cyclin T1+Cdk9)という転写伸長因子複合体を呼び寄せます。これがなければ、伸長のブロック解除(抗終結)が起こらず、転写が途中で止まってしまうため、ウイルスの増殖が著しく抑えられます。
Cdk9の活性を失わせる変異がHIV転写に及ぼす影響
- Cdk9の活性が失われると、Tat依存的な転写の伸長促進ができなくなり、HIVの遺伝子発現・複製はほぼ完全に阻害されます。
- 具体的には、Cdk9はP-TEFb複合体のキナーゼであり、RNAポリメラーゼIIや転写停止因子(NELFやDSIF)をリン酸化することで転写伸長を活性化します。Cdk9に活性がなくなると、このリン酸化が起こらず、ポリメラーゼIIは転写を途中で止めたままとなり、フルレングスのウイルスmRNAが産生されません。
- 実験的にも、Cdk9変異体(優性阻害型、ドミナントネガティブ変異)の導入によって、Tat依存的転写活性やHIV複製が大幅に抑制されることが示されています。
理由:
まとめ
- TAR変異でTatの結合ができない場合も、Cdk9活性喪失変異も、HIV転写の伸長ができず、ウイルスの発現・増殖は大きく阻害される。
- いずれも「転写伸長促進=HIVの抗終結機構」が分子レベルでも臨床応用の標的になりうる理由です。
スプライシングの際にイントロンが除去されることは、どのようにして発見されたか。エキソンーイントロン連結部の位置は、どうすれば予測できるか
スプライシングの際にイントロンが除去される現象は、1977年にウイルス遺伝子の研究を通して初めて発見されました。アデノウイルスの感染細胞内のmRNAを電子顕微鏡で観察した際、mRNA前駆体の一部がDNA上の配列とまったく一致せず、対応しない領域(後に「イントロン」と命名)が転写の過程で除去されていることが判明したのがきっかけです。この実験により、真核生物の遺伝子にはタンパク質コード領域(エキソン)と、翻訳されずスプライシングによって取り除かれる領域(イントロン)が存在することが実証され、以降スプライシング機構の研究が進展しました。
エキソン-イントロン連結部(スプライス部位)の位置は、主に以下の特徴的な塩基配列パターン(保存配列)を使って予測できます。
- イントロン5’末端(ドナーサイト)はGU(GT)で始まり、3’末端(アクセプターサイト)はAGで終わる(この配列は「GT-AGルール」と呼ばれる)。
- さらに、3’末端付近には「分岐点(ブランチポイント)」と呼ばれる保存されたA塩基の存在が重要です。
- 生物情報学的には、ゲノム配列中でこれらの保存配列パターンを探索し、より高精度な予測にはRNA-seqデータを用いた発現解析や、機械学習・隠れマルコフモデルを活用した予測方法も用いられています。
このように、イントロン除去の発見はウイルス遺伝子がきっかけであり、スプライス部位は配列モチーフなどから予測可能です。
hnRNA, snRNA, miRNA, siRNA, snoRNAの違いは何か
hnRNA(heterogeneous nuclear RNA)
- 特徴:hnRNAは「ヘテロ核RNA」とも呼ばれ、真核細胞核内で転写直後に作られる“加工前”のmRNA前駆体の総称です。エクソンとイントロンが含まれています。
- 役割:キャップ付加、スプライシング(イントロン除去)、ポリA付加などの処理を経て、成熟mRNAになります。
snRNA(small nuclear RNA)
- 特徴:snRNAは80~350塩基程度の小さなRNA分子で、細胞核内のスプライシングスペックルやカハール体に存在します。
- 役割:タンパク質と複合体(snRNP)を作り、スプライソソームの構成要素としてスプライシング(=前駆体mRNAからイントロンを除去)を制御します。
miRNA(microRNA)
- 特徴:miRNAは21〜25塩基ほどの非常に短い一本鎖RNAで、タンパク質には翻訳されません(ノンコーディングRNA)。
- 役割:mRNAの3’非翻訳領域に部分的に結合し、標的mRNAの分解や翻訳抑制を引き起こすことで遺伝子発現の負の調節を担います。細胞分化・増殖・アポトーシスなど生命現象に重要。
siRNA(small interfering RNA)
- 特徴:siRNAは21~23塩基ほどの短い二本鎖RNAです。
- 役割:RNA干渉(RNAi)の中心分子で、特定のmRNAと完全に相補的に結合することで、標的mRNAを分解・遺伝子発現を強力に抑制します。主に外来遺伝子やウイルス遺伝子の防御機構としてはたらきます。
snoRNA(small nucleolar RNA)
- 特徴:snoRNAは核小体に存在する小型ノンコーディングRNAで、多くはリボソームRNAやその他RNA遺伝子のイントロン内にコードされています。
- 役割:rRNAやsnRNAなど他のRNA分子のメチル化・シュードウリジン化など化学修飾を誘導し、リボソームの生合成やRNA機能の成熟に重要です。
まとめ表
| 種類 | 長さ/構造 | 主な存在場所 | 主な役割・機能 |
|---|---|---|---|
| hnRNA | 長い | 核 | mRNA前駆体、加工前RNA |
| snRNA | 短い | 核 | スプライソソーム構成、スプライシング制御 |
| miRNA | 短い | 主に細胞質 | mRNA分解/翻訳抑制、遺伝子発現の調節 |
| siRNA | 短い,2本鎖 | 主に細胞質 | RNA干渉による標的mRNA分解、外来遺伝子制御 |
| snoRNA | 短い | 核小体 | rRNA/snRNAなどの化学修飾誘導(メチル化・シュードウリジン化) |
それぞれ「生成時期」「存在場所」「機能」が異なることで、細胞内で多彩なRNA制御ネットワークを構築しています。
グループⅡイントロンの自己スプライシングと、スプライソソームのスプライシングとの機構上の類似点は何か。この二つの間に進化上の関係を示す証拠は何か
グループⅡイントロンの自己スプライシングとスプライソソームによるスプライシングは、一見異なる生物や分子機構による現象ですが、その化学的・構造的なメカニズムには顕著な類似性があります。また、両者の間には進化的なつながりを示唆する証拠も多く報告されています。
機構上の主な類似点
- 二段階のトランスエステル化反応
- ラリアット構造の形成
- 保存された反応部位と立体構造
- RNAによる触媒活性
進化上の関係を示す証拠
- 構造・配列の相同性
- 同様のスプライス部位配列
- タンパク質成分の進化的関連
- 遺伝学的・生化学的データ
進化的解釈
- 多くの研究者は、スプライソソーム(およびスプライソソームイントロン)はグループIIイントロンが進化して分化したものであると考えています。最初は自己スプライシング可能なリボザイム型のグループIIイントロンが原核生物や原始真核生物のゲノムに侵入・拡散し、やがてリボザイム機能を失ったイントロンと、「共通のRNAおよびタンパク質複合体によるsplicing装置(スプライソソーム)」という仕組みに再編されたと推定されます。
まとめ表
| グループⅡイントロン | スプライソソーム | |
|---|---|---|
| 反応機構 | 二段階トランスエステル化 | 二段階トランスエステル化 |
| ラリアット形成 | あり(内部A残基2′-OH使用) | あり(U2-U6 RNAが分岐点Aを協調) |
| 触媒コア | RNAによる自己スプライシング | U2/U5/U6 snRNAリボザイム活性 |
| 進化的証拠 | DV領域・内部マチュラーゼ構造体など | U6 snRNA構造・Prp8タンパク反応領域など |
| 進化仮説 | スプライソソームとイントロンの祖先 | グループIIイントロンが分断・専門化 |
研究者は、転写とRNAプロセシングはどこで起こると信じているか。これを支持する証拠は何か
現代の分子生物学研究者は、「転写(RNA合成)」と「RNAプロセシング(スプライシング、5’キャッピング、3’ポリアデニル化など)」がいずれも細胞核内で起こると信じています。
なぜそう信じられているか
- 細胞の構造観察:
真核細胞では、DNAは核という細胞内の区画に格納されており、転写酵素(RNAポリメラーゼ)やRNA加工因子も核に集中しています。このため、転写とその後続くmRNAスプライシング・修飾反応のほとんどが核内で完結します。 - プロセシングの順序と分離:
新しくできたRNA(pre-mRNA)は、核内で5’キャッピング、スプライシング、3’末端ポリアデニル化などの処理を受け、それらが完了して成熟mRNAになると初めて核膜孔を通って細胞質へ輸送され、翻訳が始まります。
一方、原核細胞(細菌など)では、転写と翻訳が細胞質でほぼ同時に起きるため、真核細胞の特徴的な仕組みとなっています。
支持する証拠
- 顕微鏡による可視化:
転写活性の高い細胞や巨大な染色体構造、例えば「ランプブラシ染色体」や「哺乳類の核内ドット」観察により、RNAポリメラーゼやスプライシング因子がDNA上の活性部位(核内)で集積している様子が直接目で確かめられています。 - 生化学的・分子生物学的実験:
細胞核ごとに抽出したmRNAを解析すると、未加工のpre-mRNAやスプライシング中間体・修飾酵素複合体などの存在が検出されます。さらに、RNA-ChIP法(クロマチン免疫沈降法)やシングルモレキュールイメージングなどの新技術により、「転写中のRNAがまさにその場でスプライシング・修飾を受けている」ことが明示されるようになりました。 - 分子のカップリングの観察:
RNAポリメラーゼIIのC末端(CTD)とmRNA修飾酵素(キャップ付加酵素、スプライシング因子、ポリアデニル化酵素など)が物理的に強固な複合体を形成しながら**転写と同時進行でプロセシングが進む(共役/co-transcriptional processing)**ことが物理化学測定や構造解析法で確認されています。 - 機能的証拠:
転写とプロセシングのどちらか一方に支障が生じると、もう一方も円滑に進まなくなる(たとえば、プロセシング不全でmRNAが核外に出られない、あるいは転写が短縮する)ことが、遺伝学的変異やノックダウン実験から明らかになっています。
まとめ:
- 転写とRNAプロセシングは、真核細胞の「核内」で主に同時進行で起こる。
- 核内で両反応が密接に連携し、mRNAの完成まで厳密に制御されていることは、顕微鏡観察・分子実験・生化学・イメージング・最新の構造研究から一貫して支持されています。
10個のエキソン、9個のイントロン、およびポリアデニル酸付加共通配列をもつ3’UTRを含む遺伝子の配列が決定されたとしょう。5番目のイントロンにもポリアデニル酸付加部位が存在した。二つのポリアデニル酸付加部位がともに使われるかどうかを調べるため、mRNAを単離したところ、筋肉組織からは長い転写産物が、他のすべての組織からは短いmRNA転写産物が見いだされたとする。これら二つの転写産物の生成に関与する機構を推測せよ。
この遺伝子のmRNAについて、筋肉組織では長い転写産物が、他の組織では短い転写産物が作られている事実から、組織特異的な「選択的ポリアデニル酸付加(alternative polyadenylation)」と「選択的スプライシング」が関与していると推測できます。
機構の推測
- 選択的ポリアデニル酸付加(alternative polyadenylation)
- mRNA前駆体の5番目のイントロンに存在するポリアデニル酸付加部位(polyAシグナル)が筋肉以外の組織で利用されることで、「短いmRNA」が生成されます。
- 一方、筋肉組織ではこのイントロン内のpolyA部位は利用されず、通常の3’UTR末端まで転写・mRNA加工が進むため、エキソン10まで含む「長いmRNA」が生成されます。
- 選択的スプライシングの関与
- 筋肉以外の組織では5番目イントロンのポリアデニル酸付加部位が使われる際に、その直前でスプライシングが中断し、MATURE mRNAはエキソン1~5までしか含まれません。
- 筋肉組織ではイントロン5のpolyA部位を飛び越してスプライシングが継続されることで、エキソン1~10および3’UTR全体がmRNAに残る形です。
- 組織特異的因子による制御
- どちらのpolyAサイトが使われるかは、筋肉細胞と他組織で発現しているRNA結合タンパク質、スプライシングファクターおよび**polyA付加因子(CPSFやCstFなど)**の濃度・活性の違いによって調節されています。
まとめ
- 短いmRNA(他組織):イントロン5内polyA部位の利用+スプライシングの終了
- 長いmRNA(筋肉):最下流(3’UTR)polyA部位の利用+全てのエキソンが含まれる
このような機構を「alternative polyadenylation(選択的ポリアデニル酸付加)」および「alternative splicing(選択的スプライシング)」と呼び、組織や発生段階に応じてタンパク質多様体や遺伝子発現調節を実現する重要な戦略です。
現象をまとめると、特定のイントロン配列に存在するpolyAシグナルの活性が組織ごとに異なり、それにより長短2種類のmRNAが生じる、ということが言えます。この制御は生理機能の多様化や組織特異性の分子基盤となっています。
RNA編集は、トリパノソーマや植物のミトコンドリア、葉緑体、そして、まれには高等真核生物で起こる基本的な現象である。RNA編集とは何か。また,ヒトのアポBの例で明らかになっている、この現象がもたらす利点は何か。
RNA編集とは何か
RNA編集(RNAエディティング)とは、DNAから転写された一次転写産物(pre-mRNAやmRNA)が、特定の塩基が他の塩基へと「書き換え」られたり、ウリジン(U)などの塩基が挿入・欠失されたりする現象です。これは、スプライシングやキャップ付加、ポリアデニル化などの一般的な転写後修飾とは異なり、「塩基配列そのものが部分的に修正される」点が特徴です。
- 塩基変換型
例:アデノシン(A)→イノシン(I)、シチジン(C)→ウリジン(U)など - 塩基挿入・欠失型
主にトリパノソーマなどの一部原生生物のミトコンドリアで観察
こうした編集によって、最終的なmRNA配列がDNAの配列と異なるものとなり、そこから作られるタンパク質の配列や性質、多様性に影響を与えます。
ヒトのアポB(Apolipoprotein B)で明らかになっている利点
ヒトでは「アポB遺伝子mRNAの編集」が有名です。アポB遺伝子からRNA編集によって2種類の異なるタンパク質(ApoB-100とApoB-48)が産生されます。
- ApoB-100:肝臓で合成され、VLDLやLDLコレステロールの主成分として働く
- ApoB-48:小腸でRNA編集により途中で終止コドンが新たに生じ、短縮型としてキロミクロンの主成分になる
この現象の分子基盤は、小腸のアポB mRNAにおいて「C(シチジン)」が「U(ウリジン)」に置換されるRNA編集が行われることにあります。これによってタンパク質の途中で「終止コドン(STOP)」が新たに生まれ、肝臓型とは異なる短いApoBタンパク質が翻訳されます。
利点・意義
- 同じ遺伝子(DNA配列)から、組織ごとに異なる機能やサイズを持つタンパク質をつくることができる
→ 遺伝子の経済的利用、多様な機能の実現 - 消化管と肝臓で異なる脂質運搬様式を発揮できる
→ 小腸では食事由来脂質を運搬(キロミクロン)、肝臓では体内脂質を運搬(VLDL/LDL)
まとめ:
RNA編集は、mRNAレベルで配列を書き換えることで1つの遺伝子から複数種類のタンパク質を作り出し、環境や組織による機能の多様化を可能にする重要な仕組みです。ヒトのアポBの例では、食物脂質の輸送と体内脂質の輸送という異なる生理的役割を持つタンパク質を作り分けることができる…これがRNA編集の「大きな利点」と言えます。
核内のタンパク質複合体は、mRNA分子を細胞質へ輸送する役割をもっている。核外輸送因子複合体を形成するタンパク質について説明せよ。また、mRNPと複合体が細胞質への方向性で動く機構の背景にある二群のタンパク質は何か。
核内で合成・修飾されたmRNAは、一連の「mRNA―タンパク質複合体(mRNP)」という形で細胞質へと輸送されます。この輸送には核外輸送因子複合体と呼ばれる特異的なタンパク質群が必要不可欠です。
核外輸送因子複合体を形成する主なタンパク質
- TAP/p15複合体(ヒトではNxf1:p15複合体)
- アダプタータンパク質
- TREX複合体(Transcription Export Complex)
mRNP複合体が細胞質方向へ動く機構と関わる二群のタンパク質
mRNAの方向性輸送を可能にするしくみの本質は、「エネルギー依存性の分子スイッチ」と「核膜孔複合体との相互作用」にあると考えられています。その中心にあるのが以下の2群のタンパク質です。
- 核外輸送受容体群(Export receptors群)
- TAP/p15など核外輸送複合体の中核となるタンパク質です。
- これらがmRNPを細胞質側へ“引っ張る”働きを担っています。
- GTP結合タンパク質(Ran GTPaseシステム)
補足
- 多くのタンパク質輸送にはこのRan-GTPサイクルが必須ですが、mRNA輸送ではTAP/p15複合体がメインで、Ranシステムに完全依存しない点で「例外」ともいえます。ただしそれでも核―細胞質間でのタンパク質の方向性輸送にはRan-GTP/GDPによる分子スイッチ機構が広く利用されています。
まとめ:
- 核外輸送因子複合体の主要構成はTAP/p15複合体およびRNA結合アダプタータンパク質。
- mRNPが細胞質方向へ「一方向」に輸送されるしくみには、(1)輸送受容体タンパク質群(TAP/p15など)、(2)GTPサイクルタンパク質群(Ranファミリー)という二群のタンパク質が協調して働いていることが基礎にあります。
RNAノックダウンは、遺伝子発現を変える一連の方法のなかでも有力な道具である。どのように遺伝子発現がノックダウンされるかを簡単に述べよ.TSC1に対する siRNAをヒト細胞に導入したときの効果を述べよ。
RNAノックダウンは、主に「RNA干渉(RNAi)」というしくみによって、目的とする遺伝子の発現を選択的に減少させる技術です。RNAiでは、標的mRNAに相補的な短い二本鎖RNA(siRNAなど)を細胞に導入すると、細胞内でこのsiRNAが「RNA誘導サイレンシング複合体(RISC)」に取り込まれます。RISCは、そのガイドとなるsiRNAの塩基配列に完全に一致するmRNAを特異的に見つけて切断・分解します。これにより、mRNAがタンパク質へ翻訳されるのを防ぎ、結果として目的遺伝子の発現量が著しく減少(部分的に抑制=ノックダウン)します。
TSC1に対するsiRNAをヒト細胞に導入したときの効果
TSC1は、細胞の増殖や分裂、分化などを制御する「mTORシグナリング経路」の重要な抑制タンパク質(ハムラートンパク質)です。TSC1に対するsiRNAをヒト細胞に導入すると、TSC1遺伝子から転写されたmRNAが分解されるため、TSC1タンパク質の量が減少します。
- 効果として、TSC1タンパク質が減ると、mTORC1シグナルの抑制が解除され、mTOR活性が増強します。
- その結果、細胞増殖やタンパク質合成が促進され、細胞周期の制御が乱れやすくなります。
- 長期的には、細胞が過剰に分裂・増殖したり、正常な分化や代謝調節に障害が生じることも示されています。
このようにして、siRNAを用いたノックダウンは、細胞内で特定遺伝子のタンパク質量を選択的かつ比較的短期間で減少させ、その生理的・分子的影響を解析するための強力な実験手法です。
Dicer活性を欠く植物では、なぜRNAウイルスの感染に対する感受性が高まるのかを推測せよ
Dicerは植物における「RNAサイレンシング機構」の中心的な酵素であり、この経路はRNAウイルスに対する主要な防御手段となっています。Dicer(DCLとも呼ばれます)は、RNAウイルスや感染過程で生じるウイルス由来の二本鎖RNA(dsRNA)を検知し、それを小さなsiRNA(small interfering RNA)断片に切断します。
Dicer活性が欠損していると何が起こるか?
- Dicerによる切断が行われないため、ウイルスRNA由来のsiRNAが作られません。
- siRNAがないと、ウイルスRNAに特異的な分解命令が出せず、RNAサイレンシング経路(RNA干渉: RNAi)が作動しなくなります。
- 結果としてウイルスRNAが細胞内で分解されず、ウイルスの複製が抑えられません。
このため、Dicer活性を持たない/欠損した植物ではRNAウイルスの感染に非常に弱くなり、ウイルスが増殖しやすくなり、感染症状が重く出現しやすくなります。
まとめ(推測の根拠)
- 植物のRNAi機構(ウイルス防御)はDicerで始まる
- Dicerがウイルス由来RNAをsiRNAに切断
- siRNAが標的ウイルスRNAを分解へ誘導(RNAサイレンシング)
- Dicerが働かない → siRNA不在 → ウイルスRNAが分解されず感染が広がる
そのため、Dicer活性を欠く植物はRNAウイルスの感染に対する感受性が高まるのです。
ある種のmRNAが,細胞内の特異的な場所に蓄積するようになっていることを示す証拠は何か
細胞内でmRNAが特定の場所に蓄積・局在する現象について、様々な実験的証拠が報告されています。主な証拠は以下のとおりです。
1. 直接観察・イメージングによる証拠
- 蛍光in situハイブリダイゼーション(FISH)やライブセルイメージングにより、特定のmRNAが細胞内の一定領域(例: 細胞突起、細胞膜直下、極性部位など)に集積している様子が可視化されています。
2. 高解像度サブセルラー解析による証拠
- サブセルラーRNAシーケンス法やプロキシミティラベリング手法により、数百~数千種類のmRNAについて、その局所分布パターンが大規模かつ詳細にマッピングされています。
3. 機能的証拠
- 局在化の制御配列やタンパク質を同定し、それらを改変(突然変異導入や欠損)した場合、mRNAが細胞内の定位置に集まらなくなり、細胞機能や分化現象に異常が生じる例が示されています。
4. 生理学的・発生学的現象
代表的な証拠まとめ:
- 蛍光染色や電子顕微鏡観察によるmRNAの偏在パターン
- 局在配列・因子の遺伝学的/分子生物学的改変効果
- サブセルラーRNAシーケンシングやイメージングによる全遺伝子規模のマッピング
- 上記の現象が細胞の分化・極性や機能の空間的制御に不可欠である確かな機能的証拠
このような多角的なエビデンスにより、「特定のmRNAが細胞内の特定の場所に蓄積・局在する」ことは分子生物学・細胞生物学において確立した現象となっています。
問題文引用元:東京化学同人 分子細胞生物学 第6版

コメント