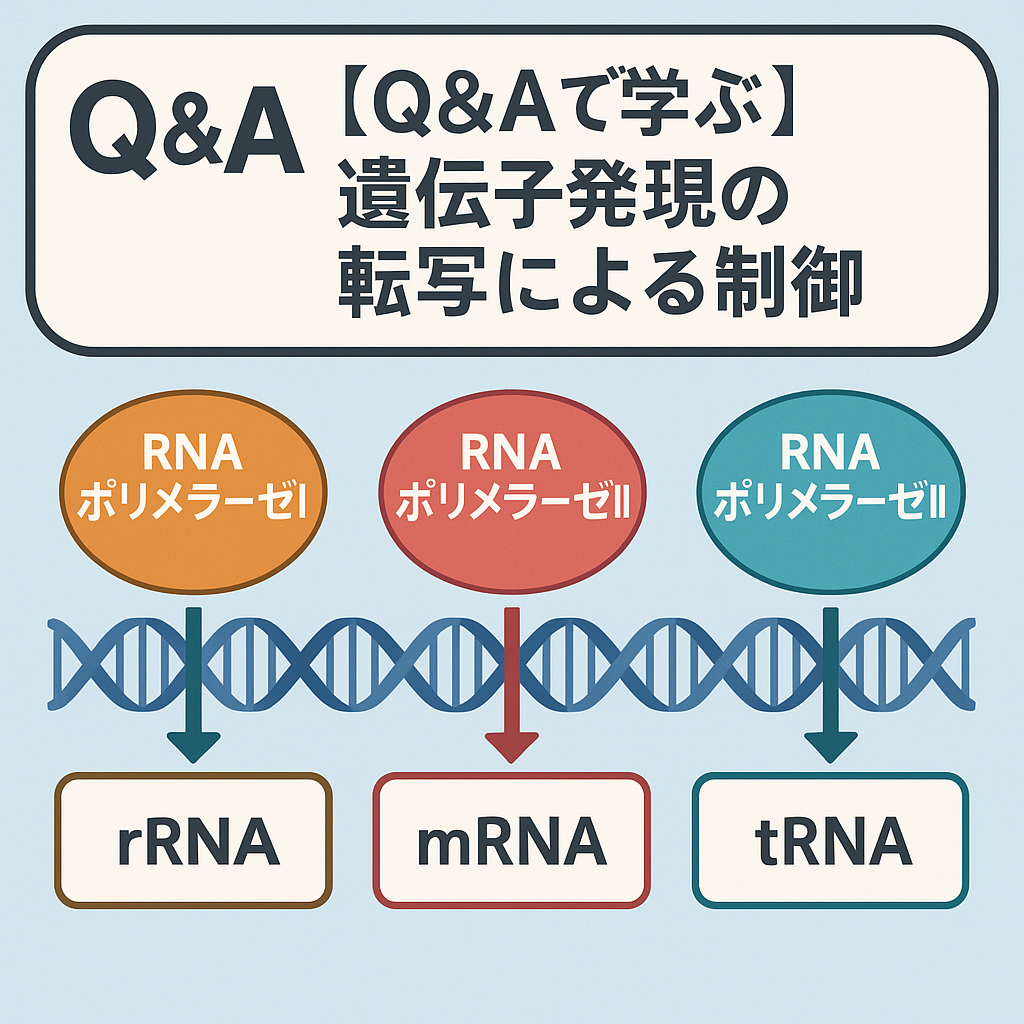
- 遺伝子は、タンパク質コード遺伝子の場合は転写されてmRNAになり、リボソーム RNA または転移RNAなどの遺伝子は転写されてそれらのRNAになる。遺伝子とは何かを定義せよ。複合転写単位から選択的プロセシングによってどのようにさまざまな mRNAと、最終的にはタンパク質ができるかを述べよ。
- ヒトゲノムの配列決定から遺伝子の構成について多くのことが明らかになった。単独の遺伝子、遺伝子ファミリー、偽遺伝子、および縦列反復遺伝子の違いを述べよ。
- ヒトゲノムの大部分は反復DNAでできている。マイクロサテライトDNAとミニサテライト DNAの違いを説明せよ。DNAフィンガープリント法で個人を特定する際に、この反復DNAはどのように利用されるか。
- 直接DNAとして新たな部位に移動、つまり転位する可動性DNA因子はDNA トランスポゾンとよばれる。細菌の挿入配列とよばれるDNA トランスポゾンの転位の機構を説明せよ.
- レトロトランスポゾンは,RNA中間体を介して転位する種類の可動性因子である。長鎖末端反復配列(LTR)を含むレトロトランスポゾンと、それを含まないレトロトランスポゾンの転位機構の違いを述べよ。
- トランスポゾンが現生生物の進化に果たしたと考えられる役割を述べよ.エキソンシャッフリングとは何か。トランスポゾンがエキソンシャッフリングの過程に果たす役割は何か。
- ミトコンドリアは独自のDNAをもっている。ミトコンドリアゲノムにコードされた遺伝子の種類を述べよ。植物,菌類,動物でミトコンドリアゲノムがどのように違うか。
- ミトコンドリアと葉緑体は核をもつ細胞に存在していた共生細菌から進化したと考えられている。この仮説を支持する実験的な証拠をまとめよ。
- なぜORF (オープンリーディングフレーム)の存在に基づいて遺伝子を配列データベースから検索することが,真核細胞ゲノムより細菌ゲノムの方が有効なのか。パラログ遺伝子とオルソログ遺伝子とは何か。ヒトは線虫に比べて遺伝子数は 1.5倍もないが(25,000対18,000),ヒトの方がはるかに複雑な生物であるということはどのように説明されるか。
- 細胞内にあるDNAはタンパク質と結合してクロマチンを形成している。ヌクレオソームとは何か、ヌクレオソームにおいてヒストンが果たす役割は何か。ヌクレオソームは、凝縮した30nm 繊維でどのように並んでいるか。
- ヒストンの翻訳後修飾は、転写されている遺伝子(ユークロマチン)と抑制されている遺伝子(ヘテロクロマチン)とでどのように異なっているか。ほとんどの真核生物では、どのようなタンパク質がヘテロクロマチンに結合しているか。これはヘテロクロマチンが染色体の領域に広がって形成されるときにどのような作用をしているか。
- 真核生物の染色体の一般的構成を述べよ。足場結合領域(SAR)もしくは基質結合領域(MAR)とよばれる構造が果たす役割とは何か。染色体構造との関係で遺伝子は主としてどこに存在するか。
- FISH は細胞遺伝学者に用いられる強力な診断方法である。FISH とは何か。ある種の遺伝病や特定の種類のがんに関連する染色体転座を調べる際にどのように用いられるかを簡単に述べよ.
- 中期染色体は特徴的な縞模様によって見分けることができる。GバンドおよびRバンドとは何か。染色体ペインティングとは何か。また,この技術はどのように利用されるか。染色体べインティングのプローブは哺乳類の染色体の進化を分析する際にどのように使われるか。
- ある生物には、多糸染色体をもつ細胞がある。多糸染色体とは何か。それはどこに存在し、どのような機能をもつか。
- 真校生物の染色体の複製と分離には、複製起点,セントロメア,およびテロメアという三つの機能要素が必要である、これら三つの要素はどのように機能するか。染色体構造を維持するうえでの、テロメラーゼの役割は何か。
遺伝子は、タンパク質コード遺伝子の場合は転写されてmRNAになり、リボソーム RNA または転移RNAなどの遺伝子は転写されてそれらのRNAになる。遺伝子とは何かを定義せよ。複合転写単位から選択的プロセシングによってどのようにさまざまな mRNAと、最終的にはタンパク質ができるかを述べよ。
遺伝子とは何か
**遺伝子**は、DNA(または一部ウイルスではRNA)分子上の特定の領域であり、遺伝的な形質を規定し、親から子へと正確に伝えられる「遺伝情報の単位」です。現代の定義では、遺伝子とは「DNA(RNAウイルスの場合はRNA)分子中の、遺伝的になんらか固有の意味を持つ、自己複製し、かつ発現して形質の発現に寄与する機能単位」とされています。各遺伝子は染色体上に配列して存在し、突然変異や遺伝的組換えによって変化することもあります。
- 遺伝子とは、生物の形質や機能を決めるための遺伝情報を持つDNAの基本単位です。
- タンパク質をコードする遺伝子は、転写されてmRNA(メッセンジャーRNA)となり、そのmRNAが翻訳されてタンパク質になります。
- 一方で、リボソームRNA(rRNA)や転移RNA(tRNA)をコードする遺伝子は、それぞれのRNA分子に転写されて直接機能を持ち、タンパク質には翻訳されません。
- 遺伝子には、プロモーター、エクソン(コード領域)、イントロン(非コード領域)、転写終結シグナルなどの構造要素が含まれます。
複合転写単位(ポリシストロン)と選択的プロセシング
複合転写単位とは
- 真核生物の中には、1つの大きな前駆体mRNA(pre-mRNA)として複数の遺伝子または遺伝子の複数エクソンを同時に転写する場合があります。
- これを「複合転写単位」や「複数のエクソンを含む転写ユニット」と呼びます。
- 複数のエクソンやイントロンを含む前駆体RNAから、細胞が様々な成熟mRNAを作り出すことが可能です。
選択的プロセシング(選択的スプライシング)
- 選択的プロセシングとは、前駆体mRNAのイントロンを除去すると同時に、**異なるエクソンの組み合わせを選択的につなぎ合わせ(スプライシング)**て複数の成熟mRNAを作る過程です。
- これにより、1つの遺伝子から複数種類のmRNAが産生され、多様なタンパク質アイソフォーム(異なるタンパク質)が合成されます。
- スプライシングのパターンによって、機能や局在の異なるタンパク質が生成されるため、遺伝子の多様性と機能範囲を拡大します。
選択的プロセシングの具体例
- エクソンのスキップ:特定のエクソンをスプライシングしないことでタンパク質の一部が欠失したバリアントができる。
- イントロンの部分的保持:一部のイントロンを残し、翻訳を調節する場合もある。
- 代替の5’または3’スプライス部位の利用:エクソンの境界をずらして長さや配列が異なるmRNAを産生する。
- 異なるプロモーターやポリA部位の利用:異なる転写開始や終結により多様なmRNAが生成される事例も含まれる。
複数のmRNAからタンパク質ができる仕組み
- 複数の成熟mRNAはそれぞれ特有のコーディング配列を持ち、リボソームで翻訳されて異なるドメイン構造や機能を持つタンパク質を生み出します。
- これにより、1つの遺伝子が多機能性を持ち、細胞の機能的多様性や調節能力が高まります。
まとめ
- 遺伝子は、タンパク質や機能性RNAをコードするDNAの基本単位であり、その産物は転写されてmRNAやrRNA、tRNAとなる。
- 複合転写単位からの転写後、選択的プロセシング(選択的スプライシング)によって異なる組み合わせのmRNAが生成されることで、最終的に多様なタンパク質が作られる。
- この仕組みは、遺伝子の多様性を増し、限られた遺伝子から多様な細胞機能を実現する重要なメカニズムである。
このように、選択的プロセシングは遺伝子情報の効率的かつ高度な活用を可能にし、生物の複雑な生命活動を支えています。
ヒトゲノムの配列決定から遺伝子の構成について多くのことが明らかになった。単独の遺伝子、遺伝子ファミリー、偽遺伝子、および縦列反復遺伝子の違いを述べよ。
単独の遺伝子
遺伝子ファミリー
- 共通の祖先遺伝子から重複・進化した、類似配列や機能をもつ複数の関連遺伝子群を指す。
- 多くは染色体上でクラスター(集団)を形成することが多いが、異なる染色体上にも分布する。
- 例:ヒトのヘモグロビンα・β鎖遺伝子ファミリーなど。
偽遺伝子
- 正常な遺伝子に類似したDNA配列だが、変異や欠損によって転写や翻訳が行われず、機能を喪失した配列。
- 多くは元の遺伝子が進化の過程でコピーや変異を重ねてできたもの。
- タンパク質をコードしないため、俗に「非機能遺伝子」とも呼ばれる。
縦列反復遺伝子
- 複数の同じ遺伝子や配列が染色体上に隣接して(連続して)反復して並ぶ形式の遺伝子。
- 主にrRNA遺伝子など、大量の発現が求められるケースで見られ、反復数は数回から数百回に及ぶこともある。
- 単位配列が頭尾連続して配列されていることが特徴。
| 種類 | 特徴 | 例 |
|---|---|---|
| 単独の遺伝子 | 染色体上で独立して存在。1種のタンパク質やRNAをコード | 血友病の原因となる特定遺伝子など |
| 遺伝子ファミリー | 共通祖先遺伝子から重複・分化した類似遺伝子群。機能や配列も類似 | ヒトヘモグロビンα・β鎖遺伝子ファミリー |
| 偽遺伝子 | 正常遺伝子配列に類似だが、機能を失った非コードDNA配列 | 進化に伴い働きを失った古い遺伝子の残骸 |
| 縦列反復遺伝子 | 染色体上で同じ遺伝子が隣接・反復して集合。大量発現や多様性確保に有利 | rRNA遺伝子群、衛星DNA、マイクロサテライト |
このように、ヒトゲノムを構成する遺伝子はさまざまなバリエーションとして存在しており、機能や進化、発現様式の違いがゲノムの多様性と生命活動の幅広さを生み出しています。
ヒトゲノムの大部分は反復DNAでできている。マイクロサテライトDNAとミニサテライト DNAの違いを説明せよ。DNAフィンガープリント法で個人を特定する際に、この反復DNAはどのように利用されるか。
マイクロサテライトDNAとミニサテライトDNAの違い
- マイクロサテライトDNA:2~6塩基対の短い配列が数回~数十回繰り返される。個人ごとの繰り返し回数が異なり、全ゲノム中に多数分布する。
- ミニサテライトDNA:10~60塩基対程度のやや長い配列が複数回(場合によっては数百回まで)繰り返され、同じく個体ごとに多型性が高い。
DNAフィンガープリント法における反復DNAの利用
- 個人識別の原理
反復DNA領域(ミニサテライトおよびマイクロサテライト)は個人ごとに繰り返し回数が異なるため、これを解析することで個人特有のDNAパターン(プロファイル)を構築できる。 - 基本的な手順
- 用途
- マイクロサテライト(STR)マーカーの強み
要点として、マイクロサテライトDNAは「非常に短い単位の繰り返し」、ミニサテライトDNAは「やや長い単位の繰り返し」であり、どちらも個人を特定するための分子マーカーとしてDNAフィンガープリント法に応用されます。
直接DNAとして新たな部位に移動、つまり転位する可動性DNA因子はDNA トランスポゾンとよばれる。細菌の挿入配列とよばれるDNA トランスポゾンの転位の機構を説明せよ.
細菌の挿入配列(IS)の転位機構
挿入配列(IS)の概要
- 挿入配列(Insertion Sequence, IS)は、細菌に存在する最もシンプルなDNAトランスポゾンです。
- ISは両端に逆向き反復配列(Inverted Repeats, IR)を持ち、その中央に転移酵素(トランスポザーゼ)遺伝子のみを含みます。
転位(転移)の流れ
- 転移酵素の発現
- IS内の転移酵素遺伝子が転写・翻訳され、トランスポザーゼが合成されます。
- 標的認識および切断
- トランスポザーゼは自分自身の両端IR配列を特異的に認識して結合し、IS配列全体をゲノムDNAから切り出します(カットアンドペースト型)またはコピーを作ります(レプリカティブ型)。
- 新たな部位への挿入
- トランスポザーゼはまた、ゲノム内の新しい標的DNA部位を探し出し、IS配列をそこで挿入します。
- 挿入の際、標的DNA配列で数塩基対が二重に複製され、**ターゲットサイト重複(Target Site Duplication, TSD)**が生じます。
- 構造変化と影響
- ISの転位により遺伝子の破壊や調節領域の変化、ゲノム構造の再編成(欠失・逆位など)が起こり得ます。
まとめ
- 細菌のISは自身の転移酵素によって、「認識–切断–新部位挿入」という一連のプロセスで直接DNAとしてゲノム内を移動します。
- この過程はトランスポザーゼの働きが不可欠で、IR配列とTSD形成を特徴としています。
- ISの転位は細菌ゲノムの可塑性、適応、進化の源となる重要な分子機構です。
レトロトランスポゾンは,RNA中間体を介して転位する種類の可動性因子である。長鎖末端反復配列(LTR)を含むレトロトランスポゾンと、それを含まないレトロトランスポゾンの転位機構の違いを述べよ。
レトロトランスポゾンの転位機構の違い
LTR型レトロトランスポゾン
- 構造的特徴
- 両末端に長鎖末端反復配列(LTR: Long Terminal Repeat)をもつ。
- 内部にgag,pol遺伝子(逆転写酵素・インテグラーゼなど)を含む。
- 転位機構
- レトロトランスポゾンDNAからmRNAが転写される。
- このmRNAは自分自身でコードする逆転写酵素によってcDNAへ逆転写される。
- 得られたcDNAはインテグラーゼにより標的ゲノムDNA内の新たな場所に挿入される。
- 挿入先でも同様のLTR配列が形成される。
- 特徴
非LTR型レトロトランスポゾン(LINE, SINE など)
- 構造的特徴
- LTR配列をもたない。
- LINEは逆転写酵素やエンドヌクレアーゼ遺伝子を持つが、SINEはそれら酵素遺伝子を持たず、自律的転位はできない。
- 転位機構
- 自身のDNA配列からmRNAが転写される。
- mRNAは細胞質で**逆転写酵素(主に自分でコードする酵素)**により逆転写される。
- このとき、標的DNA上でエンドヌクレアーゼ活性によって一本鎖切断が生じ、その3’末端が逆転写のプライマーとなる(ターゲットプライムド逆転写機構と呼ばれる)。
- 逆転写されたcDNAが直接その場に組み込まれる形(copy-and-paste)で挿入される。
- 特徴
機構の比較表
| 方式 | LTR型レトロトランスポゾン | 非LTR型レトロトランスポゾン |
|---|---|---|
| 末端配列 | LTRあり | LTRなし |
| 逆転写 | mRNA→cDNA(逆転写酵素による) | mRNA→cDNA(逆転写酵素; ターゲットプライムド型) |
| 挿入酵素 | インテグラーゼでゲノムに組込 | エンドヌクレアーゼや逆転写酵素で直接組込 |
| 転位の特徴 | LTR利用しプロモーター等の制御受ける | ランダム性が高い(挿入位置の選択性が低い) |
まとめ
LTR型はLTR配列とインテグラーゼを介した「ウイルスに類似」な転位様式、非LTR型は標的DNAで直接逆転写される独特なメカニズム(ターゲットプライムド逆転写)を持つ—この点が両者の転位機構の本質的な違いとなります。
トランスポゾンが現生生物の進化に果たしたと考えられる役割を述べよ.エキソンシャッフリングとは何か。トランスポゾンがエキソンシャッフリングの過程に果たす役割は何か。
トランスポゾンが現生生物の進化に果たした役割
- 遺伝子・ゲノム多様性の源泉
- トランスポゾン(可動性遺伝因子)は、ゲノム上を移動・増殖できるDNA配列であり、挿入・脱離・転写の活性によって遺伝子の重複、再編成、欠失などの変化を引き起こす。
- これにより、新しい遺伝子や遺伝子ファミリーの誕生や、調節配列の変動が促進され、現生生物に見られる遺伝的多様性や新規形質進化の促進に大きく寄与してきたと考えられている。
- 遺伝子発現制御への寄与
- トランスポゾン由来の配列がプロモーターやエンハンサー、サイレンサーなどの調節領域に組み込まれることで、既存遺伝子の時空間的な発現パターンにも影響を与えることがある。
- ゲノム構造の進化と適応力増進
- 染色体の再構築やゲノムサイズ増大の一因となり、特定環境・圧力下での適応進化を後押ししている。
- ヒトゲノムや様々な高等生物のゲノムでは、実に多くの部分がトランスポゾン由来である。
エキソンシャッフリングとは何か
- エキソンシャッフリング(Exon Shuffling)は、異なる遺伝子や同一遺伝子内の「エキソン(蛋白質ドメイン配列部分)」が組換えによって再配置・再編成されることをいう現象。
- この過程によって新たな遺伝子構造や新規タンパク質ドメイン構成が生じ、進化的に多様な機能や性質を持つタンパク質が誕生する。
- 代表的な効果例としては、「モジュラー構造をもつタンパク質」の出現(例:酵素、受容体の進化や新規結合ドメイン追加)があげられる。
トランスポゾンがエキソンシャッフリングに果たす役割
- エキソンの転移・再配置の仲介
- トランスポゾン(特にDNA型やレトロトランスポゾン)は、自身の構造に隣接するエキソン配列ごと“切り出し”て、ゲノムの別の位置に運ぶことがある。
- このようなプロセスで、異なる遺伝子間のエキソン再配置や挿入が起こりやすくなり、エキソンシャッフリングの分子的触媒となる。
- 組換え・再編成の活性化
- トランスポゾン挿入・挿入部位間の組換えによって、エキソン単位での遺伝子断片の新しい組み合わせが生じ、タンパク質の機能的多様化が進む。
- 進化的利点
- トランスポゾンによるエキソンシャッフリングは、従来にはなかった新たな機能ドメインや構造をもつタンパク質の爆発的な進化を可能にし、生物の適応や複雑化に大きく貢献してきた。
まとめ
トランスポゾンは現生生物の進化において「ゲノム変動」「新規機能創出」「適応進化の促進」の役割を担っており、エキソンシャッフリングの過程ではエキソン断片の組換え・再配置の“媒介者”として働くことで、多様な遺伝子とタンパク質の進化をもたらしました。
ミトコンドリアは独自のDNAをもっている。ミトコンドリアゲノムにコードされた遺伝子の種類を述べよ。植物,菌類,動物でミトコンドリアゲノムがどのように違うか。
ミトコンドリアゲノムにコードされた遺伝子の種類
ミトコンドリアゲノムは主に以下の3種類の遺伝子をコードしています。
- タンパク質コード遺伝子:
- 電子伝達系(呼吸鎖複合体)およびATP合成酵素のサブユニット(人では13種類)
- 例:NADHデヒドロゲナーゼサブユニット(ND1〜ND6等)、シトクロムb(CYTB)、シトクロム酸化酵素サブユニット(COⅠ~COⅢ)、ATP合成酵素サブユニット(ATP6、ATP8)など
- 電子伝達系(呼吸鎖複合体)およびATP合成酵素のサブユニット(人では13種類)
- リボソームRNA(rRNA)遺伝子:
- 12S rRNA、16S rRNA(人など動物では2遺伝子)
- 転移RNA(tRNA)遺伝子:
- mRNA翻訳に必要な全22種類のtRNA(人の場合)。これにより独立したタンパク合成が可能。
※ ミトコンドリアが持つタンパク質の大部分は核ゲノム由来であり、ミトコンドリア独自のDNAはごく一部の必須サブユニットや翻訳系に限定される。
植物、菌類、動物におけるミトコンドリアゲノムの違い
| 動物 | 植物 | 菌類 | |
|---|---|---|---|
| ゲノムサイズ | 10–50kb | 60kb–12Mb | 10–350kb |
| DNA構造 | 主に環状 | 主に環状(一部線状) | 環状または線状 |
| コード遺伝子数 | 非常に保守的、13タンパク質、rRNA2種、tRNA22種 | 多様、遺伝子数が多い(追加タンパク質やRNAも)、多数のイントロン | 動物と比べ多様、植物型に近い種類が多い |
| イントロン | ほとんど無し | 多数存在(主にgroup II型) | 多様(group I型・II型、種類や数が種によって大きく異なる) |
| 遺伝子順序・構成 | 高度に保守的 | 遺伝子の挿入・欠失が頻繁、組換え・再編成が多い | 配列順序が多様、組換えやイントロンによる拡大が顕著 |
| 特徴的ポイント | 遺伝子配列が比較的短く、構造がシンプル | 遺伝子含有数・ゲノムサイズが大 | 種ごとにサイズ・内容が大きく異なる、多型性が高い |
詳細な違い
- 動物
- 植物
- ゲノムサイズ・遺伝子数ともに非常に多様。反復配列や長大なイントロン、再編成(リコンビネーション)領域が多い。タンパク質コード遺伝子に加え、rRNAやtRNA、時に追加のオリジナルタンパク質もコードされることがある。
- 菌類
まとめ
- ミトコンドリアゲノムには、主に「呼吸鎖関連のタンパク質」「rRNA」「tRNA」をコードする遺伝子が存在する。
- 動物のミトコンドリアDNAは小さく単純、植物や菌類は大きく複雑で多様性が高い。イントロンや遺伝子構成の違いも大きい。
- 植物や菌類のミトコンドリアゲノムは、進化・ゲノムダイナミクスの観点から極めて多様な構造と遺伝子構成を持つ。
ミトコンドリアと葉緑体は核をもつ細胞に存在していた共生細菌から進化したと考えられている。この仮説を支持する実験的な証拠をまとめよ。
ミトコンドリアと葉緑体の起源に関する共生説(エンドシンビオント説)を支持する実験的証拠
1. 独自のDNAの保有
- ミトコンドリアと葉緑体は、核ゲノムとは別に独自の環状DNA(細菌型)を持っています。
- このDNAの塩基配列解析により、ミトコンドリアのDNAは主にαプロテオバクテリア(リケッチア類)、葉緑体のDNAはシアノバクテリアに近縁であることが判明しています。
2. 二重膜構造
- 両小器官とも二重の膜構造を持っています。外膜と内膜が異なる起源をもつことは、原始的な細菌が細胞内に取り込まれた際に形成された痕跡と考えられています。
3. 独自のリボソームとタンパク質合成
- ミトコンドリア・葉緑体内のリボソームは70S型(細菌型)であり、真核細胞質の80S型リボソームと明らかに異なります。
- 小器官内で抗生物質感受性(例:ストレプトマイシンやクロラムフェニコール)を示し、細菌のタンパク質合成系に類似しています。
4. 独立した分裂様式
- 両小器官は**二分裂法(binary fission)**で分裂増殖します。これは細菌の増殖様式と同一であり、真核細胞の有糸分裂とは独立しています。
5. 遺伝的系統解析
- ミトコンドリアDNA、葉緑体DNA、核DNAの系統樹解析では、ミトコンドリアは現存する細菌(αプロテオバクテリア)、葉緑体はシアノバクテリアに近縁であることが複数の系で明示されている。
6. 小器官由来遺伝子の核ゲノムへの移動
- 多くのミトコンドリア・葉緑体由来の遺伝子が核ゲノムに移動しており、現在もそのタンパク質の一部が小器官に輸送されて機能しています。
- この現象は、かつて独立していた細胞が長い進化の過程で一体化した名残と解釈されます。
7. 共生実験の成果
- 一部研究では、原核生物による現生真核細胞への**“人工的な共生導入系”**なども実施され、細菌が宿主細胞に組み込まれる現象が観察されています。
まとめ表
| 証拠 | 内容・意義 |
|---|---|
| 独自DNA・遺伝子系統 | 細菌に極めて近いゲノム特徴、進化系統も一致 |
| 二重膜構造 | 細胞内共生時の取り込み過程を示唆 |
| リボソーム型・タンパク質合成 | 細菌型リボソーム・タンパク質合成と同等 |
| 分裂様式 | 二分裂法による増殖 |
| 遺伝子転移・核への統合 | 機能維持のための遺伝子移動・機能分担 |
| 人工共生実験 | 実験的共生の観察 |
総合として、ミトコンドリアと葉緑体はかつて独立した細菌であり、原始真核細胞に取り込まれて二次的に進化・固定されたことを実験的に裏付ける多数の証拠が存在します。
なぜORF (オープンリーディングフレーム)の存在に基づいて遺伝子を配列データベースから検索することが,真核細胞ゲノムより細菌ゲノムの方が有効なのか。パラログ遺伝子とオルソログ遺伝子とは何か。ヒトは線虫に比べて遺伝子数は 1.5倍もないが(25,000対18,000),ヒトの方がはるかに複雑な生物であるということはどのように説明されるか。
ORF(オープンリーディングフレーム)による遺伝子検索が細菌ゲノムで有効な理由
- 細菌(原核生物)ゲノムでは、遺伝子がほぼ連続して配置されており、多くはイントロンを持ちません。また、ほとんどのタンパク質コード遺伝子が一つの大きなORF(長いコドン連続)として明瞭に現れます。
- このため、配列をスキャンして長いORFを見つけるだけで、ほとんどの遺伝子を漏れなく検出することが可能です。
- 一方、真核生物では、エクソン(コード領域)とイントロン(非コード領域)が複雑に入り組んでおり、1つの遺伝子でも複数の短いORFが分断されて存在します。また、ノンコーディングRNAや擬似遺伝子も多数含まれるため、単純なORF探索だけでは正確な遺伝子同定が困難であり、追加の情報(スプライシング予測・配列アノテーションなど)が不可欠です。
パラログ遺伝子とオルソログ遺伝子の違い
| 種類 | 定義 |
|---|---|
| パラログ(paralog) | 同一生物種内で、遺伝子重複によって生じた、異なる機能(または類似機能)をもつ遺伝子。 |
| オルソログ(ortholog) | 別々の生物種に存在し、**種分岐(共通祖先)**を起点に分かれた機能的に相同な遺伝子。 |
ヒトと線虫で遺伝子数が近いのにヒトがはるかに複雑な理由
- 遺伝子数(ヒト:約25,000、線虫:約18,000)はそれほど大きく違いませんが、生物の複雑性は単純な遺伝子数だけでは説明できません。
- 複雑さの主な要因
- タンパク質ファミリーとドメインの多様性
ヒトでは、1つの遺伝子から多様なスプライシング(選択的スプライシング)によって異なるタンパク質バリアントが多数作られます。さらに、多くの異なるタンパク質ファミリーや機能ドメインが存在し、タンパク質間のネットワークも高度です。 - 転写・翻訳後修飾と遺伝子調節機構
ヒトなど高等動物では、遺伝子発現の調節(エピジェネティクス、ノンコーディングRNAなど)が非常に複雑で、状況に応じた多様な細胞タイプや機能分化が可能です。 - 細胞の種類・組織の多様さ
複雑な生物は、より多くの細胞型や組織を持ち、発生や機能が高度に分化しています。線虫は細胞数も約1,000個程度でほぼ決まっていますが、ヒトは数百種類、数兆個の細胞から成ります。
- タンパク質ファミリーとドメインの多様性
- まとめ
参考:
細胞内にあるDNAはタンパク質と結合してクロマチンを形成している。ヌクレオソームとは何か、ヌクレオソームにおいてヒストンが果たす役割は何か。ヌクレオソームは、凝縮した30nm 繊維でどのように並んでいるか。
ヌクレオソームとは何か
- **ヌクレオソーム**は、真核細胞の核内におけるクロマチン(DNAとタンパク質複合体)の基本構造単位です。
- 約146塩基対のDNAが、8分子からなるヒストンコア(H2A, H2B, H3, H4 各2個ずつ)の周囲を1.65回巻き付くことで形成されます。
- さらに、ヒストンH1がDNAの出入り口部分に結合することで、ヌクレオソーム同士の連結や高次構造化が進みます。
- ヌクレオソームは「ビーズ・オン・ア・ストリング(紐に通したビーズ)」状に、連続して細胞核内のDNA上に並んでいます。
ヌクレオソームにおけるヒストンの役割
- **ヒストン**は、DNAを安定してパッケージングするためのコアとなる塩基性タンパク質です。
- コアヒストン(H2A, H2B, H3, H4)が八量体を形成し、DNAを巻き付けて物理的に保護・安定化します。
- これにより、DNAが核内で無秩序に広がることなく、コンパクトにまとめられます。
- ヒストンは、クロマチンの構造変換や転写制御にも関与し、ヒストン修飾(メチル化、アセチル化など)を通じて遺伝子発現を調節する重要な機能も持っています。
- ヒストンH1は、ヌクレオソームどうしの連結や高次構造(30nm繊維)形成に不可欠で、ヌクレオソーム同士をさらに集積させます。
ヌクレオソームの30nm繊維での配列
- ヌクレオソームが「ビーズ・オン・ア・ストリング」状に列を成した後、ヒストンH1の作用などにより30nm繊維へと高次に凝縮されます。
- 30nm繊維では、ヌクレオソームが以下のように規則正しく配列されます:
- ソレノイドモデル(らせん型)
ヌクレオソームが6個ずつらせん状の輪(ソレノイド)を形成し、30nmの太さで繊維状に並ぶとされます。 - ジグザグモデル
ヌクレオソームがジグザグに交互に並び、リンクDNAがより直線的に伸びる構造も提唱されています。
- ソレノイドモデル(らせん型)
- この30nm繊維の集積がさらに上位のクロマチンループや染色体構造へとつながります。
| 構造段階 | 概要 |
|---|---|
| ヌクレオソーム | コアヒストン+DNAの基本単位 |
| 10nm繊維 | 「ビーズ・オン・ア・ストリング」状配列 |
| 30nm繊維 | 複数ヌクレオソームが凝縮、らせん構造等 |
ポイント:
- ヌクレオソームはDNAのコンパクト化と遺伝子制御の基本単位である。
- ヒストンはDNAの巻き付け・高次凝縮・遺伝子制御に必須。
- 30nm繊維では、ヌクレオソームが規則正しく集積し、効率的なDNAの収納・管理に寄与している。
ヒストンの翻訳後修飾は、転写されている遺伝子(ユークロマチン)と抑制されている遺伝子(ヘテロクロマチン)とでどのように異なっているか。ほとんどの真核生物では、どのようなタンパク質がヘテロクロマチンに結合しているか。これはヘテロクロマチンが染色体の領域に広がって形成されるときにどのような作用をしているか。
ヒストンの翻訳後修飾とクロマチン状態の違い
ユークロマチン(転写活性遺伝子)の特徴
- 主なヒストン修飾
- 高頻度のヒストンアセチル化(特にH3およびH4のリジン残基)
→ クロマチン構造が緩み、転写因子やRNAポリメラーゼがアクセスしやすくなる。 - 一部の部位ではヒストンH3のリジン4(H3K4)のメチル化、H3K36のメチル化なども“活性マーク”として知られる。
- 高頻度のヒストンアセチル化(特にH3およびH4のリジン残基)
- 効果
ヘテロクロマチン(転写抑制領域)の特徴
- 主なヒストン修飾
- 低頻度のアセチル化(ヒストンの脱アセチル化)
- リジン9(H3K9)やリジン27(H3K27)のメチル化が顕著(例:H3K9me2/3,H3K27me3など)
- 効果
ヘテロクロマチンに結合するタンパク質
- ほとんどの真核生物に存在する代表的なヘテロクロマチン結合タンパク質はHP1(Heterochromatin Protein 1)ファミリー。
ヘテロクロマチンの領域拡大とタンパク質の役割
- 領域拡大(スプレッディング)のメカニズム
- 拡大の機能的意義
- 広範な領域で遺伝子発現を抑制し、「サイレンシングドメイン」を形成。
- 反復配列や転移因子の沈静化、染色体安定性の確保にも重要。
- 境界領域(バリアエレメント)がないと、周囲のユークロマチン領域にも抑制が及ぶことがある。
まとめ表
| クロマチン状態 | 主要修飾 | 主な結合タンパク質 | 転写状態 | スプレッディング |
|---|---|---|---|---|
| ユークロマチン | アセチル化多, H3K4me等 | 予備的(少ない) | 活性化 | なし |
| ヘテロクロマチン | 脱アセチル化, H3K9me多 | HP1ファミリー等 | 強い抑制 | HP1による拡大可 |
ポイント
- ユークロマチンは“アセチル化多+メチル化少”で開いた構造、ヘテロクロマチンは“脱アセチル化+H3K9メチル化”で閉じた構造。
- HP1などのタンパク質がH3K9meに結合し、メチル化の正のフィードバックでヘテロクロマチンが領域として広がる。
真核生物の染色体の一般的構成を述べよ。足場結合領域(SAR)もしくは基質結合領域(MAR)とよばれる構造が果たす役割とは何か。染色体構造との関係で遺伝子は主としてどこに存在するか。
真核生物の染色体の一般的構成
- 一本の染色体はDNA二重らせん分子と多種多様なタンパク質(主にヒストン)から構成され、核内でクロマチンとして存在します。
- 主な構造要素は以下のとおりです。
| 構造レベル | 主な特徴・内容 |
|---|---|
| DNA | 約数千万~数十億塩基対の線状二本鎖分子 |
| ヌクレオソーム | DNAがヒストン八量体に約146塩基対分巻き付く基本構造単位 |
| 10nm繊維 | ヌクレオソームが「ビーズ・オン・ア・ストリング状」に連なる |
| 30nm繊維 | ヌクレオソームがさらに螺旋化、凝縮した状態 |
| ループドメイン構造 | 30nm繊維が非ヒストンタンパク質の“足場”に結合して形成 |
| メタフェーズ染色体 | 細胞分裂中の高度に折りたたまれた状態 |
- 染色体には機能領域としてセントロメア(動原体)、テロメア(末端構造)、そして多数の遺伝子領域が存在します。
足場結合領域(SAR)/基質結合領域(MAR)の役割
- SAR(Scaffold Attachment Region)/MAR(Matrix Attachment Region)は、クロマチンDNA配列のうち、核内の非ヒストンタンパク質で構成される“足場構造”やマトリックスに特異的に結合する領域です。
- 主な役割:
- クロマチン高次構造の組織化
- 30nm繊維が“足場”や“マトリックス”に結合することで、大きなループ構造(ループドメイン)を形成します。
- 1つのループドメインには複数の遺伝子が含まれ、これにより遺伝子発現や複製、修復などが局所的に調節しやすくなります。
- 染色体領域の機能的区画化と安定化
- ループの境界にSAR/MARが機能することで、隣接する領域のエピジェネティック修飾が互いに干渉しにくくなります。
- サイレンシングや活性化ドメインの“絶縁体”として働き、染色体構造と遺伝子機能の両方を安定化します。
- クロマチン高次構造の組織化
染色体構造との関係で遺伝子はどこに主に存在するか
- 遺伝子は主にクロマチンの「ループドメイン」内、特にユークロマチンと呼ばれる“ほどけやすく転写活性な領域”に多数存在します。
- 染色体の異常に凝縮したヘテロクロマチン領域(セントロメア近傍やテロメア付近など)には遺伝子が少なく、反復配列やジャンクDNAが多く含まれています。
- 一方、クロマチン構造が開いているユークロマチン領域にこそ、細胞で必要な多くの遺伝子が集積しています。
ループ構造の“内部”や“先端”付近が転写活性に富み、SAR/MARによる固定化によって隣接する領域から機能的に分離されています。
まとめ
真核生物の染色体はヒストンとDNAおよび足場タンパク質の複合体で、SAR/MARがクロマチンの高次折り畳み・領域機能の区画化に中心的役割を果たしています。遺伝子は主にユークロマチン領域のループドメイン内部に多く配置され、細胞機能の維持に必要な遺伝子発現が効率的に制御されています。
FISH は細胞遺伝学者に用いられる強力な診断方法である。FISH とは何か。ある種の遺伝病や特定の種類のがんに関連する染色体転座を調べる際にどのように用いられるかを簡単に述べよ.
FISHとは何か
- **FISH(Fluorescence In Situ Hybridization:蛍光 in situ ハイブリダイゼーション)**は、染色体やDNA配列を直接可視化し、特定の遺伝子、染色体領域、あるいは染色体異常を高感度かつ特異的に検出する分子細胞遺伝学的手法です。
- 原理は、蛍光色素で標識したDNAプローブ(相補的配列)を固定化した細胞や染色体上にハイブリダイズ(結合)させることで、目的の配列の位置や数、構造異常を蛍光顕微鏡により直接観察します。
染色体転座の診断とFISHの利用方法
- 染色体転座とは、異なる染色体間でDNA断片が交換される構造異常の一種で、遺伝病や多くのがん(白血病、リンパ腫など)で特徴的に認められます。
- FISHでは、転座が疑われる二つの染色体上のそれぞれの領域に特異的な異なる色の蛍光プローブを設計・使用します。
- 正常な細胞では各プローブはそれぞれ分離した場所にシグナルが見られます。
- 転座が起こると、異なる染色体由来のプローブシグナルが「並ぶ」「重なる」「融合(融合シグナル)」して観察され、転座の存在を直接証明できます。
- 例えば、慢性骨髄性白血病(CML)でみられるt(9;22)(フィラデルフィア染色体)の検出や、特定の白血病やリンパ腫における診断・病型鑑別などに幅広く利用されています。
FISHの診断応用のポイント
- わずか数個の細胞や細胞分裂像(中期染色体)から高精度・短時間で染色体異常の同定が可能。
- 組織切片、血液標本、間期細胞など多様な検体に適用できる。
- 遺伝病: 21トリソミー(ダウン症)、微小欠失症候群など染色体構造異常の迅速診断。
- 腫瘍・がん: 染色体転座、増幅、欠失の同定で分子病型分類や治療方針決定に直結。
まとめとして、FISHは遺伝子や染色体異常の“その場検出”を可能にし、遺伝病やがんの診断・分類・予後予測に不可欠な細胞遺伝学的ツールです。
中期染色体は特徴的な縞模様によって見分けることができる。GバンドおよびRバンドとは何か。染色体ペインティングとは何か。また,この技術はどのように利用されるか。染色体べインティングのプローブは哺乳類の染色体の進化を分析する際にどのように使われるか。
GバンドおよびRバンドとは何か
Gバンド(Giemsaバンド)
- Gバンドは、染色体解析で最も広く使われているバンディング法です。中期染色体(凝縮した染色体)をGiemsa染色で染めることで、各染色体に特徴的な縞模様(バンド模様)を生じさせます。
- 原理: 染色体を酵素処理(通常トリプシン)した後、Giemsa染色を施します。AT塩基に富み遺伝子が少ないヘテロクロマチン領域は濃く(暗色バンド)染まり、GC塩基に富み遺伝子の多いユークロマチン領域は淡く(明色バンド)なります。
- このバンドパターンは各染色体ごとに固有で、各バンドに番号が振られ、染色体異常(欠失・転座など)の診断や染色体の識別に使用されます。
Rバンド(リバースバンド)
- **Rバンド**は、Gバンドの「逆」の染色パターンを作るバンディング法です。
- 原理: 染色体を高温の緩衝液で前処理したのち、Giemsaなどで染色します。これにより、GC塩基の多い領域が暗色バンドとなり、AT塩基の多い領域が明色バンドとなります(Gバンドとは反対)。染色体の末端領域や細かい領域の観察で特に有効です。
染色体ペインティングとは何か
- **染色体ペインティング(chromosome painting)**は、**蛍光in situハイブリダイゼーション(FISH)**を利用し、特定染色体やその一部を蛍光色素で可視化する分子細胞遺伝学的な技術です。
- 方法: 染色体特異的なDNA配列(プローブ)を蛍光色素で標識し、細胞内または染色中のDNAにハイブリダイゼーションさせます。目的の染色体だけが鮮やかな色で「塗り分け(ペイント)」されるので、「染色体ペインティング」と呼ばれます。
- 複数のプローブと異なる蛍光色素を組み合わせることで、1細胞中の複数染色体を同時に色分け・識別することも可能です。
染色体ペインティングの利用方法
- 染色体異常・転座の検出
Gバンディングで難しい微小な転座・再編成・欠失や重複も、ペインティング法によって明瞭に可視化できます。これにより、がん細胞や遺伝病などの染色体構造異常の診断、研究に活用されます。 - “カラー核型”での判別や種間比較
人やマウスなどでは「マルチカラーFISH」(M-FISH)を使い、全染色体を色分けしたカラー核型を作成。これにより、複雑な再編成や微細構造を一目で判別できます。 - 研究応用の例
・特定遺伝子や領域の物理的位置探査
・生殖細胞や腫瘍の微小モザイク異常の解析
・染色体分配や不均等分裂の解析
染色体ペインティングプローブによる哺乳類染色体進化の分析
- 進化比較の手法
ヒトなどの1本の染色体に対応するDNAプローブ(「ペイント」)をサル、マウス、ゾウ、ギボンなど他種の染色体にFISHでハイブリダイズさせると、**進化的に保存された領域(シンテニ―)**や、転座・融合・分裂等の染色体再編成がどこで生じたかが可視化できます。 - 進化の解明への実際例
例えば、人染色体2号相当の領域が他のサルでは分裂・転座して複数染色体に分かれている、といった進化過程における染色体構造変化の比較研究が可能となっています。 - 比較染色体地図の構築
多種間でペインティングを相互に行い、どの領域が共通祖先に由来するか、どこで再編成が起きているかを明らかにし、「哺乳類の染色体進化史」をたどる解析に広く利用されています。
まとめ
- **Gバンド**はAT-rich領域が暗色、RバンドはGC-rich領域が暗色となる染色体のバンディング法。
- **染色体ペインティング**はFISHによる染色体分画法で、疾病診断や染色体進化の比較に不可欠な技術となっている。
- 哺乳類進化解析では、「プローブ」を使い染色体構造の保存・再編成を種間で比較し、進化パターンや祖先染色体の復元など大きな発見がもたらされている。
| 技術 | 原理・特徴 | 主な用途・意義 |
|---|---|---|
| Gバンディング | Giemsa染色、AT-richが暗色 | 染色体の特定・異常検出 |
| Rバンディング | 熱・染色でGC-richが暗色 | 末端解析・構造異常検出 |
| 染色体ペインティング | FISHによる染色体塗り分け | 異常検出、進化比較、核型判定 |
ある生物には、多糸染色体をもつ細胞がある。多糸染色体とは何か。それはどこに存在し、どのような機能をもつか。
多糸染色体とは何か
多糸染色体(polytene chromosome)は、数千本ものDNA鎖が束ねられた巨大な染色体です。これは、細胞分裂を伴わずにDNA複製が何度も繰り返された結果、姉妹染色分体が分離せず一つの巨大な構造体となったものです。通常よりもはるかに大きく、光学顕微鏡でもそのバンド模様が明瞭に観察できるのが大きな特徴です。
多糸染色体の存在場所
多糸染色体は、さまざまな生物の特定組織や細胞で見られますが、代表的なのは次のようなものです。
- 昆虫(特にハエ目):
ショウジョウバエやユスリカなどの幼虫の唾液腺、分泌組織、マルピーギ管などで顕著に観察されます。これらの染色体はしばしば「唾液腺染色体」とも呼ばれます。 - その他:
原生動物の一部、節足動物の体細胞、特定の植物(豆類の未熟種子や胚柄の細胞など)、哺乳類の胎盤栄養膜細胞でも報告があります。
多糸染色体の機能
多糸染色体には以下のような重要な機能があります。
- 高レベルな遺伝子発現
DNA配列が多数コピーになっているため、限られた時間内に大量のRNAやタンパク質を産生できます。例えばショウジョウバエ幼虫の唾液腺では、蛹化直前に大量の粘着タンパク質を合成する必要があるため、唾液腺細胞でこの構造が発達しています。 - 遺伝学・染色体地図作成への利用
バンド模様が明瞭なため、染色体異常の検出や遺伝子のマッピングなど、細胞遺伝学的解析や分類研究に応用されてきました。 - 活発な転写・染色体構造変化の観察
バンドの一部が膨らんで「パフ(puff)」と呼ばれる構造が形成されることがあり、これは遺伝子の活発な転写が起こっている領域を示します。パフの発現はホルモン刺激や発生段階に応じて変化し、転写活性の直接的な観察対象となります。 - 細胞の体積増大や機能分担
多糸染色体をもつ細胞は核自体も大きくなり、細胞の機能的拡張や分化に適応しています。
| 特徴 | 内容 |
|---|---|
| 正体 | 数千本のDNA鎖が束状につながった巨大な染色体 |
| 代表的な存在場所 | 昆虫(唾液腺)、一部の植物細胞、原生動物、哺乳類 |
| 主な機能 | 大量の遺伝子発現、遺伝学的地図作製、転写観察 |
多糸染色体は、高度な機能分化と効率的な大量遺伝子産物の必要性に応じて発達した特殊な染色体です。
真校生物の染色体の複製と分離には、複製起点,セントロメア,およびテロメアという三つの機能要素が必要である、これら三つの要素はどのように機能するか。染色体構造を維持するうえでの、テロメラーゼの役割は何か。
真核生物染色体の三大機能要素の役割
1. 複製起点(replication origin)
- 役割
複製起点は、DNA複製が始まる特定配列であり、DNA合成酵素複合体(複製開始因子)が最初に集合して二重らせんを開き、新しいDNA鎖の合成がスタートする場所です。 - 機能
染色体全体が忠実かつ効率的に複製されるために、染色体上に多数の複製起点が配置されており、複製タイミングや反応の協調制御にも寄与します。
2. セントロメア(centromere)
- 役割
セントロメアは染色体の中央部に存在し、細胞分裂時に動原体(キネトコア)が形成される領域です。 - 機能
細胞分裂時の紡錘糸がこの領域に結合し、姉妹染色分体が正確にそれぞれの娘細胞へ分配されることを保証します。
セントロメアがない場合、染色体は片方の娘細胞に偏ってしまい、遺伝情報の正確な分配ができなくなります。
3. テロメア(telomere)
- 役割
テロメアは染色体の両末端に存在する特殊な反復配列(ヒトではTTAGGGの繰り返し)で、染色体末端の安定性・保護を担います。 - 機能
通常のDNA複製では「末端複製問題」(DNAポリメラーゼが末端の全てを複製できず、徐々に末端が短くなる)が生じるため、テロメアがバッファーとなって遺伝情報の消失を防ぎます。
また、末端部が「傷んだDNA」や「異常な二重鎖切断」として細胞が誤認するのを防ぎ、染色体端の融合や不安定化を抑制します。
テロメラーゼの役割
- テロメラーゼ(telomerase)は、テロメア配列を新たに合成・伸長する酵素複合体です。
- 主な機能
- 核酸逆転写酵素活性(RNA鋳型を利用し、テロメア末端に反復配列を追加)
- 体細胞では一般的に活性が低く、細胞分裂ごとにテロメアが短縮するが、幹細胞や生殖細胞(および多くのがん細胞)ではテロメラーゼが高活性で、テロメアの長さを維持または回復する
- 意義
- 染色体の末端を長期間維持し、染色体構造や遺伝子の安定性、細胞寿命の確保に不可欠
- テロメラーゼが働かない場合、テロメア短縮が進むと細胞老化やゲノム不安定化(染色体消失や融合など)を引き起こす
| 機能要素 | 主な役割 |
|---|---|
| 複製起点 | DNA複製の開始点として働き、全染色体が複製される体制を作る |
| セントロメア | 分裂時の紡錘糸結合・染色体分配の司令塔となり、遺伝情報を正確に伝える |
| テロメア | 染色体両端の保護・複製限界の緩和、融合防止 |
| テロメラーゼ | テロメアを再構築し、染色体末端と細胞の長寿命化を支える |

コメント