- 大腸菌をグルコースを含む培地からラクトースを含む培地に移したときに、lacオペロンで起こる現象を分子レベルで述べよ。
- 遊離リン酸の濃度は、いくつかの大腸菌遺伝子の転写に影響を与える。その機構を述べよ。
- RNA ポリメラーゼI 、IIおよびIIIによってそれぞれどのような種類の遺伝子が転写されるか。ある遺伝子がRNA ポリメラーゼIIによって転写されるかどうかを決める実験を考案せよ。
- RNA ポリメラーゼIIの最大サブユニットのCTDでは,複数のセリン残基がリン酸化される。どのような条件によって、このCTDのリン酸化状態と非リン酸化状態が導かれるのか。
- TATAボックス、イニシエーター、およびCpGアイランドが共通にもつ作用は何か。そのなかで最初に同定されたのはどれか。また、それはなぜか。
- 遺伝子のプロモーター近くの領域にあるDNA 調節エレメントの位置を同定するために用いられる方法を述べよ
- プロモーター近位エレメントと、離れたエンハンサーの違いは何か。
- 遺伝子の調節領域内にあるDNA結合タンパク質の結合部位を同定するために用いられる方法を述べよ
- 転写アクチベーターおよびリプレッサーのタンパク質構造上の特徴を述べよ
- ウィルムス腫瘍の患者ではEGR1遺伝子の転写に何が起こっているのか、また、それはなぜか。
- CREB と核内受容体を例に、これらの転写因子がコアクチベーターに結合したときに起こる構造変化を比較し、その違いを述べよ。
- RNAポリメラーゼIIのプロモーターに結合するポリメラーゼ以外の基本転写因子は何か。それらがin vitroで結合する順序はどのようになっているか。“開いた”転写開始複合体が形成されたときにDNAに起こる構造変化は何か。
- 組換えタンパク質を酵母に発現させることは,ヒトに使用するために新たな薬剤を生産するバイオテクノロジー企業にとって重要な手段である。ある研究者は、新しい遺伝子を酵母で発現させようと考えて,遺伝子Xを酵母ゲノムのテロメアの近くに組込んだ。この方法で遺伝子Xをうまく発現させることができるか否か。また,その理由を述べよ。この実験において、ヒストン H3またはH4の尾部に突然変異をもつ酵母の系統を使った場合には、実験結果は違うだろうか。
- いま STICKYとよばれる新しいタンパク質を単離したとしよう。他の既知のタンパク質との比較から,STICKYにはbHLHドメインとSin3相互作用ドメインが含まれると予測された。これからSTICKYの機能を予想し、STICKYの機能におけるこれらのドメインの重要性について理論的に予測せよ。
- 酵母ツーハイブリッドシステムは、既知のタンパク質やタンパク質ドメインに相互作用するタンパク質を同定するための有力な分子遺伝学的方法である。ここで、グルココルチコイド受容体(GR)が活性化ドメイン、DNA 結合ドメイン、リガンド結合能をもつ第二の活性化ドメインをもつモジュールタンパク質である証拠があったとしよう。さらに解析した結果,下垂体細胞では、このタンパク質はリガンドであるホルモンがない場合には細胞質にとどまっていることがわかったことから、これは他の阻害タンパク質と結合しているだろうと考察した。どのように酵母ツーハイブリッドシステムを用いて、GRが相互作用するタンパク質を同定することができるかを述べよ。また、GRにおいて、阻害タンパク質を特異的に結合するドメインを同定するにはどのようにすればよいか.
- 熱ショック遺伝子にコードされるタンパク質には、過酷な条件から他のタンパク質をすばやく守る働きをするものがある。このような遺伝子を制御するために進化した機構を述べよ。
大腸菌をグルコースを含む培地からラクトースを含む培地に移したときに、lacオペロンで起こる現象を分子レベルで述べよ。
1. 基本状態(グルコース存在下)
- グルコースが豊富な培地では、エネルギー源としてグルコースが最優先して利用されます。
- このとき、大腸菌がラクトースを分解する必要はないため、lacオペロンは転写抑制状態にあります。
- 主な要因として「カタボライト抑制(グルコース効果)」が働きます。
- グルコース存在下では、細胞内のcAMP濃度が低下し、CAP(catabolite activator protein)-cAMP複合体がlacプロモーターに結合できません。
- また、lacオペロンのリプレッサー(LacIタンパク質)がオペレーター配列に結合し、RNAポリメラーゼの転写開始を物理的にブロックします。
2. 培地をラクトースへ切り替えた直後の変化
(1) cAMP濃度の上昇とCAP活性化
- グルコースが枯渇し、エネルギー源がラクトースになると、細胞内のcAMP量が上昇します。
- cAMPはCAPと結合し、「CAP-cAMP複合体」を形成します。
- この複合体がlacオペロンのプロモーター上流のCAP結合部位へ結合し、RNAポリメラーゼの転写開始効率を高める働きをします(正の制御)。
(2) ラクトース(または誘導体アロラクトース)によるリプレッサーの阻害
- 細胞外のラクトースが細胞内へ取り込まれると、その一部はアロラクトース(ラクトースの誘導体)へ代謝されます。
- アロラクトースはlacリプレッサータンパク質に結合し、立体構造を変化させることで、リプレッサーがオペレーターから脱離します(負の制御が解除)。
(3) lacオペロンの転写開始
- CAP-cAMP複合体がプロモーターに結合し、リプレッサーがオペレーターから離れたことでRNAポリメラーゼがlacオペロンプロモーター部位に結合しやすくなります。
- その結果、lacZ、lacY、lacA遺伝子(β-ガラクトシダーゼ等)からなるmRNAが転写されます。
(4) タンパク質の合成とラクトース代謝
- 転写されたmRNAはリボソームにより翻訳され、以下の酵素が合成されます。
- β-ガラクトシダーゼ(lacZ産物):ラクトースをグルコースとガラクトースに分解
- ラクトースパーミアーゼ(lacY産物):ラクトースの細胞内への取り込み促進
- トランスアセチラーゼ(lacA産物)
- 結果として、大腸菌はラクトースを新たなエネルギー源として利用できるようになります。
まとめの流れ
| 状態 | 分子レベルの反応 |
|---|---|
| グルコース存在下 | cAMP↓、CAP非活性化、リプレッサーがオペレーターへ結合→転写停止 |
| ラクトース存在下 | cAMP↑、CAP-cAMP複合体形成・プロモーター結合、リプレッサー阻害解除→転写促進 |
ポイント
- グルコース→ラクトース切替時、lacオペロンは「負の制御(リプレッサー)、正の制御(CAP)」が連動的に解除され、ラクトース利用遺伝子が効率的に発現するという精緻な分子調節ネットワークが働きます。
遊離リン酸の濃度は、いくつかの大腸菌遺伝子の転写に影響を与える。その機構を述べよ。
大腸菌では、細胞外や細胞内の無機リン酸(遊離リン酸)濃度が特定の遺伝子群の発現をダイナミックに制御しています。中心的なのが「リン酸レギュロン(Pho regulon)」の制御です。
1. PhoR-PhoB 二成分調節系
- 大腸菌は、「PhoR」(膜貫通型センサーキナーゼ)と「PhoB」(応答調節タンパク質:転写因子)から成る二成分調節系によりリン酸濃度の変動を感知し、遺伝子発現を制御します。
低リン酸濃度下(リン酸欠乏時)の応答
- シグナルの認識と伝達
- 外部のリン酸濃度が低いと、PhoRが自己リン酸化し、そのリン酸基をPhoBに転移してPhoBをリン酸化(活性化)します。
- 転写活性化
- リン酸化されたPhoBは、リン酸レギュロン遺伝子(リン酸輸送体や代謝酵素など)のプロモーター領域にある「Phoボックス(Pho box)」に結合します。
- これにより標的遺伝子の転写が促進され、細胞は不足したリン酸を効率良く取り込もうとします。
高リン酸濃度下の応答
- シグナルの遮断
- 細胞がリン酸に十分満たされると、PstSCAB/PhoU複合体(高親和性リン酸輸送体システム)がPhoRに抑制シグナルを伝達。
- PhoRはホスファターゼ活性を示してPhoBからリン酸基を除去し、PhoBが非活性化されます。
- 転写抑制
- 非リン酸化PhoBはDNAに結合できず、リン酸レギュロン遺伝子の転写は低下します。
2. Pho regulonに含まれる遺伝子
- 主要ターゲットには、リン酸取得に関与する高親和性輸送体(pstSCABオペロン)、非有機リン酸エステラーゼ(phoAなど)、リン酸枯渇時の代謝酵素群などが含まれます。
3. 分子レベルのまとめ
- 低リン酸→PhoR活性化→PhoBリン酸化→標的遺伝子の転写活性化
- 高リン酸→PstSCAB/PhoU複合体の作用→PhoRホスファターゼ活性→PhoB非リン酸化→転写抑制
この調節は、大腸菌が環境のリン酸濃度に迅速かつ柔軟に応答し、資源の効率的な取り込みと節約を両立する分子機構です。
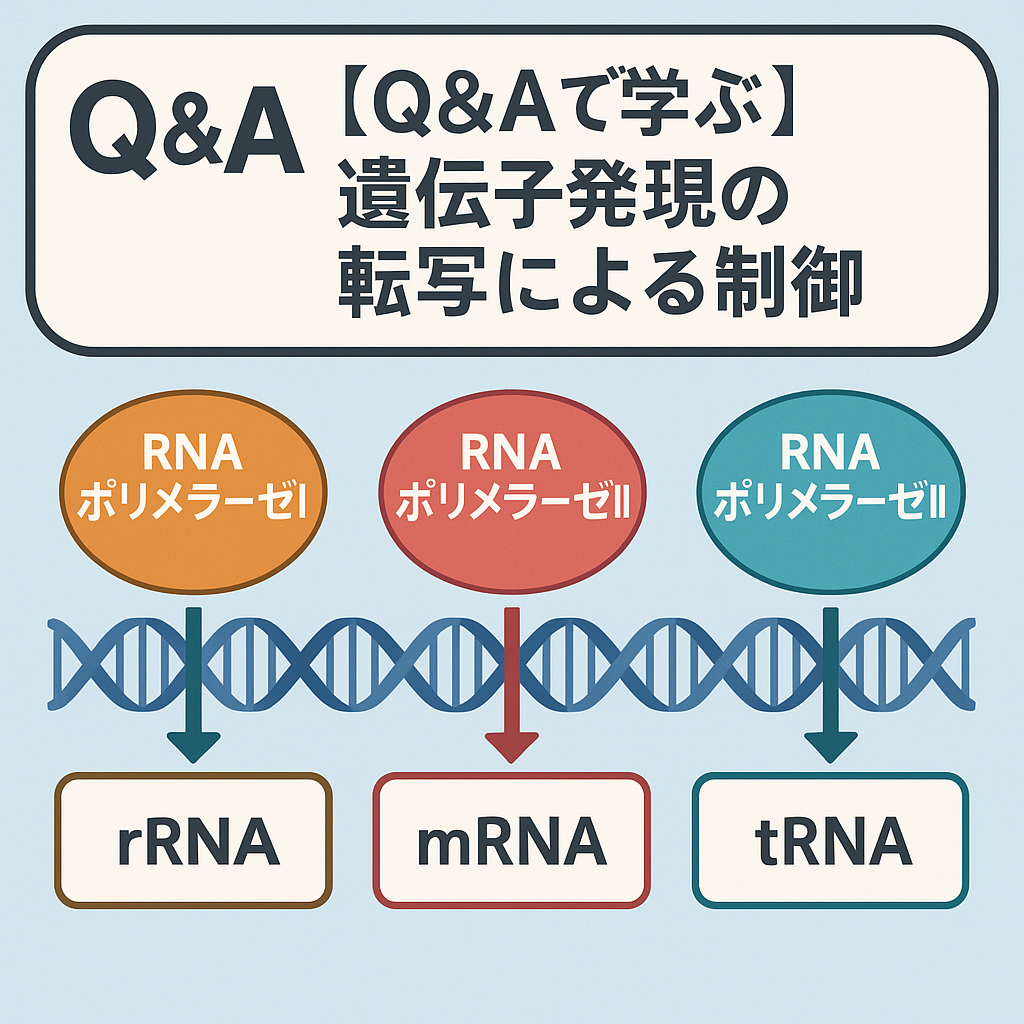
RNA ポリメラーゼI 、IIおよびIIIによってそれぞれどのような種類の遺伝子が転写されるか。ある遺伝子がRNA ポリメラーゼIIによって転写されるかどうかを決める実験を考案せよ。
各RNAポリメラーゼが転写する遺伝子の種類
詳細
- Pol Iは核小体でリボソーム構成RNAを大量生産する専用ポリメラーゼで、真核細胞の全RNA合成量の大部分を占めます。
- Pol IIはmRNAの他、転写調節やスプライシングに関わるsplicing snRNA(U1, U2, U4, U5)、miRNAなども転写します。
- Pol IIIはtRNAや5S rRNA、各種小型機能RNAなど、非翻訳性の「ハウスキーピングRNA」等の転写を担います。
RNAポリメラーゼII依存的転写かどうか調べる実験の例
- α-アマニチンによる阻害実験
- クロマチン免疫沈降法(ChIP)
- RNAポリメラーゼIIに特異的な抗体を用いて、目的遺伝子のプロモーター領域または遺伝子体への結合を検出する。
- ChIPでPol II結合が確認できれば、その遺伝子はPol IIによって転写されていると考えられる。
- in vitro転写系解析
- 精製したPol I, II, IIIそれぞれと目的遺伝子配列を用い、どのポリメラーゼで転写産物が得られるか直接確認する方法もある。
まとめ
- Pol Iは大型リボソームRNA、Pol IIは全mRNAや多くの小型調節RNA、Pol IIIはtRNAや5S rRNAなどを転写します。
- Pol II依存性の決定実験はα-アマニチン阻害、ChIP解析、in vitro系など、複数手法で高い確証を得るのが一般的です.
RNA ポリメラーゼIIの最大サブユニットのCTDでは,複数のセリン残基がリン酸化される。どのような条件によって、このCTDのリン酸化状態と非リン酸化状態が導かれるのか。
CTD(C-末端ドメイン)とセリン残基のリン酸化
- RNAポリメラーゼIIの最大サブユニット(Rpb1)は、C末端に「CTD(C-terminal domain)」と呼ばれる反復配列(ヒトで52回反復、配列はYSPTSPS)を持っています。
- このCTD配列のセリン残基(主にSer2, Ser5, Ser7)がリン酸化・脱リン酸化を繰り返すことで、転写のさまざまな段階や条件に応じて機能的な変化が生じます。
CTDの非リン酸化状態とその条件
- 初期状態(転写開始前)
- Pol IIが転写開始点(プロモーター付近)にリクルートされる時点では、CTDはほぼ非リン酸化(unphosphorylated)な状態です。
- TFIID、Mediator複合体、転写開始因子(TFII系)などが集まって「転写開始前複合体(pre-initiation complex)」を形成しやすいのは、この非リン酸化CTDへの結合が効率的に行えるからです。
- 条件
- 転写前プロモーターDNAでの組立や転写因子の結合時、CTDは非リン酸化を保ちます。
- 転写終了後、特定のホスファターゼ(例:FCP1)によってCTDは再び非リン酸化されて「リサイクル」されます。
CTDのリン酸化状態とその条件
- 転写開始〜伸長段階
- TFIIH複合体に含まれるCDK7(Kin28など)がCTDのSer5残基を主にリン酸化します。
- このリン酸化により、転写開始複合体が崩れてPol IIは「転写伸長」へ進む準備が整います。
- Ser5リン酸化は、キャッピング酵素やスプライシング因子のリクルートにも重要で、転写初期イベントの目印となります。
- 転写伸長の途中では、次にCDK9(P-TEFb複合体)などがSer2残基をリン酸化します。
- Ser2リン酸化はエクソン–イントロンのスプライシングやポリA付加因子の結合と連動し、転写伸長とmRNAプロセシングが協調して進みます。
- TFIIH複合体に含まれるCDK7(Kin28など)がCTDのSer5残基を主にリン酸化します。
- 条件
- CDK7/TFIIHが活性化されている状態(転写開始の際)でSer5リン酸化が起こる。
- P-TEFbの活性化(転写伸長期)がSer2リン酸化を促進する。
- クロマチン構造や細胞シグナル、発現プロモーターごとの調節により、CTDリン酸化パターンは動的に制御されます。
CTDリン酸化と非リン酸化のまとめ
| 状態 | 主なリン酸化残基 | 機能的意義 | 条件・制御因子 |
|---|---|---|---|
| 非リン酸化 | なし | 転写開始複合体組立、因子リクルート | 転写開始前、転写終了直後、ホスファターゼ |
| Ser5リン酸化 | Ser5 | 5’キャッピング、転写開始の開放 | TFIIH(CDK7)、転写開始付近 |
| Ser2リン酸化 | Ser2 | 伸長、スプライシング、ポリA付加 | P-TEFb(CDK9)、転写伸長域 |
参考ポイント
- CTDのリン酸化/脱リン酸化は、転写開始、伸長、終結、mRNAプロセッシングなど多段階で動的に変化し、専用のキナーゼやホスファターゼで厳密に調節されています。
- 非リン酸化→Ser5リン酸化→Ser2リン酸化→非リン酸化という繰り返しが、Pol IIサイクルの大枠です。
まとめ
RNAポリメラーゼIIのCTDは、セルリン残基のリン酸化状態によって転写制御のさまざまな段階を規定し、リン酸化状態は転写開始、転写伸長、細胞内シグナル、特定キナーゼやホスファターゼの活性など、多数の要因によってダイナミックに導かれています。
TATAボックス、イニシエーター、およびCpGアイランドが共通にもつ作用は何か。そのなかで最初に同定されたのはどれか。また、それはなぜか。
共通にもつ作用
- TATAボックス、イニシエーター(Inr)、CpGアイランドはいずれも「遺伝子の転写開始に関与する配列(プロモーター領域内の構造要素)」です。
- これらはRNAポリメラーゼⅡなど転写装置がDNA上の遺伝子を認識し、転写を開始する位置や頻度に影響を与えるという共通した役割をもっています。
それぞれの概要
- TATAボックス:典型的なプロモーター配列。転写開始点の上流約25塩基に多く見つかり、転写装置の集まりやすさを高めます。
- イニシエーター(Inr):TATAボックスがないプロモーターで見られることが多く、転写開始点を正確に指定します。
- CpGアイランド:シトシンとグアニンが連続する配列が多い領域で、特に家系性・構成的(ハウスキーピング)遺伝子のプロモーター付近に多く、転写の活性化や制御に関与します。
最初に同定されたのはどれか
- TATAボックスが最初に同定されました。
その理由
- TATAボックスは、酵母やウイルス、ヒトなど様々な生物の転写開始部位を比較することで、特定のDNA配列が繰り返し観察されたため、1970年代に比較的早く発見されました。
- 比較的配列が明確かつ繰り返し現れやすいため、他の構造に比べて発見が容易でした。
- 一方、イニシエーターやCpGアイランドは配列のバリエーションが多く、機能的特定や発見がやや遅れました。
まとめ
| 名前 | 主な共通作用 | 最初の発見 | 発見理由 |
|---|---|---|---|
| TATAボックス | 転写開始調節(プロモーター配列) | 最初 | 配列が単純で解析しやすく、繰り返し発見されたため |
| イニシエーター | 転写開始点の特定(TATAなしでも機能) | TATA次第 | 配列の多様性があるため発見がやや遅れた |
| CpGアイランド | 転写制御、発現維持(構成遺伝子プロモーター) | TATA次第 | 分布が複雑で解析が進んだ後に明確に定義されたため |
これらは「転写の開始位置や制御」に関連して働く重要なDNA配列要素であり、特にTATAボックスが歴史的にも最初に発見されました。
遺伝子のプロモーター近くの領域にあるDNA 調節エレメントの位置を同定するために用いられる方法を述べよ
- DNase I フットプリンティング法
DNase I酵素でDNAを部分分解し、タンパク質が結合している領域(調節エレメント)は分解されず“足跡”として検出される。このことから転写因子などが結合する調節領域を高精度で特定できる。 - ゲルシフトアッセイ(EMSA)
標識したDNAとタンパク質を反応させて形成される複合体のゲル中での移動度の変化を観察することで、DNA上の調節エレメントへのタンパク質結合を検出し、結合部位を推定できる。 - クロマチン免疫沈降法(ChIP)
生体内でDNAとタンパク質の結合をクロスリンクし、特定の転写因子やヒストン修飾を抗体で回収、その結合部位をDNAシークエンスやPCRなどで同定する手法。近年ではChIP-seqとして全ゲノム規模で調節エレメントを特定することができる。 - リポーターアッセイ
調節エレメントと考えられる配列断片をリポーター遺伝子の上流に連結し、細胞または動物に導入して発現の変化を見ることで、調節領域として機能するか、またその位置情報を調べる。 - ATAC-seq(染色質アクセシビリティアッセイ)
トランスポザーゼを使って開いた染色質(タンパク質が結合していない領域)に特異的にタグを付け、配列解析することで、プロモーター近傍を含む調節エリアを全ゲノム規模で迅速に同定できる。
いずれの方法も、それぞれの特性や解析目的に応じて使い分けられており、複数の手法を組み合わせてDNA調節エレメントの正確な位置の同定や機能の解析が進められている。
プロモーター近位エレメントと、離れたエンハンサーの違いは何か。
プロモーター近位エレメントとエンハンサーはいずれも遺伝子の発現調節に関わるDNA配列ですが、位置や働き方に明確な違いがあります。
主な違い
| 項目 | プロモーター近位エレメント | エンハンサー |
|---|---|---|
| 位置 | プロモーターのすぐ近く(通常は数十~数百塩基対上流) | 遺伝子から数千~数万塩基対離れても存在する。上流・下流・イントロン内など多様 |
| 働き方 | 主にプロモーターの活性化や基礎的な転写調節 | 転写活性を大きく増強する。細胞種や発生段階特異性、環境応答性が強い |
| 依存性 | 対象のプロモーター配列と密接に協調する | 距離や方向性にあまり依存せず、ループを形成してプロモーターに作用する |
| 認識される因子 | 一般的な転写因子 | 細胞種特異的な転写因子など多様 |
| 機能の具体例 | TATAボックス周辺のGCボックスやCAATボックスなど | βグロビン遺伝子のLCRや多くの発現促進領域 |
詳細解説
- プロモーター近位エレメントは「プロモーター」のすぐ隣に位置し、多くは転写開始点の100~200塩基対上流に存在します。これらは転写因子の結合サイトとなって、基本的な転写活性の調整や、遺伝子がどのくらい発現するかの“ベースライン”となる部分を担います。
- エンハンサーは「遺伝子から物理的にかなり離れた位置」に存在しても機能するのが特徴です。DNAループ構造によってプロモーターへ物理的に“接近”し、あるいはクロマチン構造を変化させることで、標的遺伝子の転写を大幅に活性化します。エンハンサーの働きは、細胞の種類や発生段階、外部刺激によって大きく変わるため、非常に精密な遺伝子制御に関わっています。
まとめ
- 位置の違い:プロモーター近位エレメントは遺伝子のすぐ傍、エンハンサーは遠く離れた場所にも存在。
- 役割の違い:前者は基礎的な転写促進、後者は発現の大幅な増強や細胞・時期特異的制御を担う。
- 働き方:プロモーター近位エレメントは通常その付近のプロモーターのみに作用、エンハンサーは遠隔でも標的遺伝子へ情報を伝達できる。
遺伝子の調節領域内にあるDNA結合タンパク質の結合部位を同定するために用いられる方法を述べよ
遺伝子の調節領域(プロモーターやエンハンサーなど)にDNA結合タンパク質がどの部位に結合しているかを調べるには、以下のような方法が広く用いられています。
主な同定方法
- クロマチン免疫沈降法(ChIP)/ChIP-seq
生細胞や組織内でDNAとタンパク質を架橋し、特定のDNA結合タンパク質(転写因子など)を抗体で免疫沈降させます。その後、回収したDNA断片の配列をPCRや次世代シークエンサー(ChIP-seq)で解析することで、ゲノム上の結合部位を網羅的に同定できます。 - DNase Iフットプリンティング法
DNA断片をDNase I酵素で軽く消化し、タンパク質が結合している部分は切断から守られ“フットプリント”として検出されます。このフットプリント領域を解析することで、保護されている=タンパク質が結合している部位を特定できます。 - DMSフットプリンティング法(ケミカルフットプリンティング)
ジメチル硫酸(DMS)などの化学修飾剤を使い、DNA中の保護された領域=タンパク質結合部位を検出します。DMS-seqのように次世代シークエンシングと組み合わせることで網羅的な結合解析も可能です。 - CUT&Tag法
Cleavage Under Targets and Tagmentationの略で、抗体で特定のDNA結合タンパク質を認識し、その部位でDNAを酵素により切断・断片化します。断片化されたDNAを解析することで精密に結合部位を同定できます。従来のChIP-seqより細胞数が少なくても可能で、近年注目の新しい手法です。 - ゲルシフトアッセイ(EMSA)
DNA断片とタンパク質を混合し、電気泳動の移動度変化から結合の有無や部位を解析します。より細かな変異導入と組み合わせて特定塩基配列への結合性も調べられます。 - DNAアレイ・マイクロアレイ法
種々の塩基配列を持つDNA断片を基板上に並べ、標識したDNA結合タンパク質や細胞核抽出液を反応させ、どの配列に結合するかを網羅的に調べる手法です。
これらの方法は、それぞれ解析のスケールや分解能、必要なサンプル量などの特徴があります。目的や材料に応じて適切な手法が選ばれています。
転写アクチベーターおよびリプレッサーのタンパク質構造上の特徴を述べよ
転写アクチベーターとリプレッサーは、遺伝子発現の調節にかかわる調節タンパク質であり、それぞれ特有の構造的特徴を持っています。
転写アクチベーター(Transcription Activator)の構造的特徴
- DNA結合ドメイン(DBD: DNA Binding Domain)
- 特定の塩基配列(エンハンサーやプロモーター)を認識して結合する部分。
- よくみられるモチーフ:ヘリックス-ターン-ヘリックス、ジンクフィンガー、ロイシンジッパー、ヘリックス-ループ-ヘリックスなど。
- 活性化ドメイン(Activation Domain)
- 基本転写因子やRNAポリメラーゼなど転写装置と相互作用し、転写活性を高める領域。
- 酸性アミノ酸リッチ領域(例:グルタミン酸やアスパラギン酸が多い)、プロリンリッチ、グルタミンリッチ、セリン/スレオニンリッチなどのタイプがある。
- 分子間相互作用ドメイン
- 他の転写因子、コアクチベーター、または調節タンパク質と相互作用するための領域。
まとめ(アクチベーターの代表的な構造要素)
| ドメイン名 | 主な機能 | 構造例 |
|---|---|---|
| DNA結合ドメイン | 特定配列への結合 | ジンクフィンガー、HTHなど |
| 活性化ドメイン | 転写装置との相互作用・転写促進 | 酸性リッチ、プロリンリッチ等 |
| 相互作用ドメイン | 他のタンパク質との結合 | 多様 |
転写リプレッサー(Transcription Repressor)の構造的特徴
- DNA結合ドメイン(DBD)
- アクチベーター同様、特定配列(しばしばオペレーター領域)に結合する領域。
- 同じく各種モチーフ(ヘリックス-ターン-ヘリックスなど)を持つものが多い。
- リプレッション(抑制)ドメイン
- 転写を妨げるさまざまな因子(コリプレッサー、クロマチン修飾酵素など)と結合し、転写の抑制を担う領域。
- シノグルタミンリッチ、メチオニンリッチドメイン、ユビキチン化関連部位など多様。
- コリプレッサー結合ドメイン
- コリプレッサーやヒストン修飾因子と結合し、転写装置の活性を低下させる。
まとめ(リプレッサーの代表的な構造要素)
| ドメイン名 | 主な機能 | 構造例 |
|---|---|---|
| DNA結合ドメイン | 特定配列への結合 | ヘリックス-ターン-ヘリックス等 |
| リプレッションドメイン | 抑制因子やクロマチン修飾因子との結合 | シノグルタミンリッチ等 |
| コリプレッサー結合部 | コリプレッサーとの相互作用 | 多様 |
ポイント
- アクチベーターもリプレッサーも「DNA結合ドメイン」と「調節ドメイン」を持っているが、調節ドメイン(活性化or抑制)の機能と相互作用相手が異なる点が大きな特徴です。
- 多くの因子は複数のドメインから構成されており、同じタンパク質内で複数の役割を果たす場合もあります。
ウィルムス腫瘍の患者ではEGR1遺伝子の転写に何が起こっているのか、また、それはなぜか。
ウィルムス腫瘍(Wilms腫瘍)は主に小児に発生する腎臓の悪性腫瘍で、このがんの発生にはWT1(Wilms Tumor 1)遺伝子とEGR1(Early Growth Response 1)遺伝子の相互作用が深く関わっています。
EGR1遺伝子の転写に何が起こっているか
それはなぜか
- 主な理由は、WT1遺伝子産物(WT1タンパク質)がEGR1の働きを抑制するからです。
具体的な分子機構
- WT1とEGR1は類似した部位に結合し、WT1が結合することでEGR1の結合や活性化作用を競合的に阻害します。
- そのため、WT1遺伝子の高発現や異常が見られるウィルムス腫瘍では、EGR1の標的遺伝子の発現およびEGR1自身の発現が低下しやすくなります。
まとめ表
| 遺伝子/タンパク質 | ウィルムス腫瘍での挙動 | 結果・影響 |
|---|---|---|
| WT1 | 過剰発現 | EGR1の作用を阻害し、EGR1標的遺伝子の発現を低下させる |
| EGR1 | 発現の低下・ばらつきが生じる | 腫瘍抑制や正常な発生分化の制御が働きにくくなる |
ウィルムス腫瘍では、WT1タンパク質の過剰発現によりEGR1遺伝子の転写活性が抑えられ、その結果としてEGR1が本来持つ腫瘍抑制機能や分化調節機能が十分に発揮されなくなることが、腫瘍の発生や進行に関与していると考えられています。
CREB と核内受容体を例に、これらの転写因子がコアクチベーターに結合したときに起こる構造変化を比較し、その違いを述べよ。
CREBと核内受容体のコアクチベーター結合時の構造変化の比較
転写因子であるCREB(cAMP Response Element-Binding protein)と核内受容体は、いずれもコアクチベーターとの結合によって転写活性を調節しますが、その結合に伴う構造変化には特徴的な違いがあります。
CREBのコアクチベーター結合時の構造変化
- 基本構造
CREBは基本ロイシンジッパー(bZIP)ドメインを持ち、DNA結合および二量体化を行います。 - コアクチベーターとの相互作用部位
主にCREB結合タンパク質(CBP)やCREB共活性化因子(CRTC)などがコアクチベーターとして機能します。 - 構造変化の特徴
- コアクチベーターのKIXドメインは、CREBの活性化ドメイン(リン酸化されたセリンを含む)と結合することで、柔軟なループ部分やC末端のαヘリックスの形成・安定化を誘導します。
- この結合により、CBPのKIXドメインの構造が部分的に秩序化され、安定した複合体が形成されます。
- CREB自身も活性化ドメインの一部が構造をとり、コアクチベーターとの相互作用面が拡大されます。
- 結果
この構造的秩序化により、転写複合体の形成が促進され、下流遺伝子の転写活性が増強されます。
核内受容体のコアクチベーター結合時の構造変化
- 基本構造
核内受容体はリガンド結合ドメイン(LBD)を持ち、ホルモンなどのリガンド結合により構造が変化し転写調節機能を持ちます。 - コアクチベーターとの相互作用部位
主にp160ファミリーのコアクチベーター(SRC-1など)がLBDの特定の溝(ヒドロフォビックグルーブ)にあるLxxLLモチーフを介して結合します。 - 構造変化の特徴
- リガンドがLBDに結合するとLBD内のαヘリックス(特にヘリックス12)がリポジショニングし、コアクチベーターペプチドの結合ための結合ポケットが形成されます。
- この変化はLBDのC末端付近の構造を大きく変え、コアクチベーターのLxxLLモチーフが溝にフィットするように誘導されます。
- リガンド依存的なアロステリック移行により、核内受容体は高親和性でコアクチベーターを捕捉できる状態になります。
- さらに、結合後にはホモまたはヘテロ二量体のLBD間の相互作用や構造非対称性も誘導され、転写活性の調節が促進されます。
- 結果
構造変化によりコアクチベーターが安定的に結合し、その招集によってヒストン修飾やクロマチン再構築が進み、標的遺伝子の転写が活性化されます。
CREBと核内受容体の構造変化の主な違い
| 項目 | CREB | 核内受容体 |
|---|---|---|
| コアクチベーター結合部位 | KIXドメイン(CBP) | LBDのヒドロフォビックグルーブ |
| 主要な構造変化の箇所 | CBPのKIXドメインの局所的秩序化 | LBDのヘリックス12のリポジショニング |
| 構造変化の性質 | 柔軟なループとC末端ヘリックスの形成・安定化 | αヘリックスの大きな位置変化と結合ポケット形成 |
| リガンド依存性 | 主にリン酸化修飾に依存 | ホルモンなどリガンド結合に強く依存 |
| 立体構造の変化の広がり | コアクチベーター側の局所秩序化が中心 | 受容体側の大規模コンフォメーション変化 |
まとめ
- CREBの場合、コアクチベーター(CBPなど)結合により、コアクチベーターの一部領域が秩序を持つようになるとともに、CREBの活性化ドメインが整列し、安定的な複合体形成を実現する。この構造変化は比較的局所的で柔軟性を伴う。
- 核内受容体の場合、リガンド結合によって受容体自身のLBDの立体構造が大きく変化し、コアクチベーター結合のためのポケットが形成される。この変化は受容体内で比較的大規模で明確なコンフォメーションの移行を伴う。
- 両者ともコアクチベーター結合によって転写活性化が促進されるが、構造変化の機構および依存因子で大きく異なり、それぞれの機能的役割に最適化されていると言える。
以上の構造的特徴と違いは、転写制御の分子メカニズムを理解するうえで重要な知見となっています。
RNAポリメラーゼIIのプロモーターに結合するポリメラーゼ以外の基本転写因子は何か。それらがin vitroで結合する順序はどのようになっているか。“開いた”転写開始複合体が形成されたときにDNAに起こる構造変化は何か。
RNAポリメラーゼIIプロモーターに結合する基本転写因子
RNAポリメラーゼIIによる転写開始には、以下の「基本転写因子(General Transcription Factors, GTFs)」が必要です:
- TFIID(TATAボックス結合タンパク質TBP+TBP関連因子TAFs)
- TFIIA
- TFIIB
- TFIIF
- TFIIE
- TFIIH
これらはRNAポリメラーゼII(Pol II)以外に、プロモーター上で必須の因子です。
in vitroでの結合順序
in vitro再構成系で観察されるプロモーターへの結合順序は、次のようになります:
- TFIIDが最初にプロモーター上のTATAボックスに結合する(これによってプロモーターが認識される)。
- TFIIAが結合し、TFIIDによるDNA結合を安定化する。
- TFIIBがTFIID複合体に結合し、RNAポリメラーゼIIとTFIIFのリクルートメントの足場となる。
- RNAポリメラーゼIIとTFIIFが同時にプロモーターにリクルートされる。
- TFIIEが複合体に結合する。
- TFIIHが最後に結合し、プロモーター上に完全なプレイニシエーション・コンプレックス(PIC)が形成される。
この順序は「組み立て型(stepwise assembly)」と呼ばれています。
“開いた”転写開始複合体が形成されたときのDNAの構造変化
転写開始複合体が「閉じた複合体」から「開いた複合体(open complex)」に移行すると、以下のようなDNA構造変化が生じます:
- DNAの二重らせんが転写開始点付近(通常 -9 ~ +2 付近)でほどけて、一本鎖の「転写バブル」が形成される。
- この“転写バブル”内で、鋳型鎖(テンプレートストランド)がRNA合成の鋳型として解放され、非鋳型鎖(センス鎖)から離れる。
- このDNAの解離はTFIIHのヘリカーゼ活性によって促進され、エネルギー(ATP)を用いて行われる。
- 結果として、RNAポリメラーゼIIの活性部位に鋳型DNA鎖が正しく配置され、RNA合成の初期伸長が可能となる。
この際、プロモーターDNAは局所的に「融解」し、10塩基対以上の領域が一本鎖化されます。
まとめ表
| 因子名 | 役割/特徴 | プロモーターへの結合順 |
|---|---|---|
| TFIID | プロモーター認識、TBPでTATAボックス結合 | 1 |
| TFIIA | TFIIDの結合安定化 | 2 |
| TFIIB | Pol IIリクルートメントの足場 | 3 |
| TFIIF | Pol IIと一緒にリクルート | 4 |
| TFIIE | TFIIHのリクルートおよび活性調節 | 5 |
| TFIIH | DNAの融解(開環)/ヘリカーゼ活性 | 6 |
開いた転写開始複合体(オープンコンプレックス)ではDNAが部分的に一本鎖化され、転写のスタートが切れる構造的準備が整います。
組換えタンパク質を酵母に発現させることは,ヒトに使用するために新たな薬剤を生産するバイオテクノロジー企業にとって重要な手段である。ある研究者は、新しい遺伝子を酵母で発現させようと考えて,遺伝子Xを酵母ゲノムのテロメアの近くに組込んだ。この方法で遺伝子Xをうまく発現させることができるか否か。また,その理由を述べよ。この実験において、ヒストン H3またはH4の尾部に突然変異をもつ酵母の系統を使った場合には、実験結果は違うだろうか。
テロメア近傍への遺伝子組込みと発現の可否
- 遺伝子Xを酵母ゲノムのテロメア近く(サブテロメア領域)に組込んだ場合、遺伝子Xは一般的にうまく発現しない可能性が高いです。
テロメア周辺領域(サイレンスドクロマチン、サブテロメア領域)は「遺伝子サイレンシング」が起こりやすく、外来遺伝子や本来存在する遺伝子の転写が抑制される傾向があります。この現象は「テロメアポジションエフェクト(telomere position effect, TPE)」と呼ばれます。 - 理由として、テロメア周辺のクロマチンは強く凝縮されており、転写因子やRNAポリメラーゼなどの転写装置がDNAへアクセスしにくくなっているためです。
そのため、たとえ強力なプロモーターを利用しても、テロメア近傍に組換え遺伝子を組み入れた場合、期待したレベルでの発現が得られない場合が多くなります。
ヒストンH3またはH4の尾部に突然変異をもつ系統を使った場合の影響
- ヒストンH3やH4の尾部は、クロマチンの構造制御や遺伝子サイレンシングに深く関与しています。
- ヒストン尾部に変異が入ることで、サイレンシング関連タンパク質(例:Sirタンパク質など)の結合が弱くなったり、クロマチン構造の凝縮が緩む場合があります。
- その結果、サブテロメア/テロメア近傍における遺伝子の“サイレンシング効果”が低下し、遺伝子Xが本来よりも高く発現できるようになる可能性があります。
実際に、ヒストンH3やH4尾部に機能喪失変異が入った酵母では、通常サイレントな状態にある遺伝子の発現が解除される現象が報告されています。
まとめ
| 条件 | 遺伝子Xの発現 | 理由 |
|---|---|---|
| テロメア近傍(野生型ヒストン) | 低い/発現しにくい | テロメアポジションエフェクトによりサイレンシングされる |
| ヒストンH3またはH4尾部変異株のテロメア近傍 | 高まる可能性あり/発現しやすい | サイレンシングが緩和されて転写装置がアクセスしやすくなる |
- 結論として、テロメア周辺に遺伝子Xを導入すると発現が抑制されやすいが、ヒストンH3/H4尾部に変異を持つ酵母を使うとサイレンシングが緩み、遺伝子Xの発現が向上する可能性があります。
いま STICKYとよばれる新しいタンパク質を単離したとしよう。他の既知のタンパク質との比較から,STICKYにはbHLHドメインとSin3相互作用ドメインが含まれると予測された。これからSTICKYの機能を予想し、STICKYの機能におけるこれらのドメインの重要性について理論的に予測せよ。
機能予測
STICKYはbHLH(basic Helix-Loop-Helix)ドメインとSin3相互作用ドメインの両方を持つことから、転写因子としての機能と、転写抑制複合体との結合による制御機能の両面を持つと考えられます。
1. bHLHドメインの役割
- DNA結合とダイマー形成機能:bHLHドメインはDNA上の特定配列(E-box: CANNTG)を認識し結合するため、STICKYが標的遺伝子のプロモーターやエンハンサー領域に直接結合できる可能性が高いです。
- ダイマー化:bHLHドメインは同じbHLHドメインを持つ他のタンパク質とホモダイマーまたはヘテロダイマーを形成し、複合体として遺伝子発現を促進あるいは抑制します。
- 細胞分化や増殖制御:bHLHドメインを持つ因子は神経系や筋肉、血液細胞の発生・分化など、多様な細胞運命決定や発現調節の主要な役割を果たします。
2. Sin3相互作用ドメインの役割
- 転写抑制複合体のリクルート:Sin3はヒストン脱アセチル化酵素(HDAC)を含む転写抑制複合体の足場(scaffold)として働きます。Sin3相互作用ドメインを持つタンパク質はSin3複合体に結合し、標的遺伝子周辺のクロマチン環境を変化させて遺伝子発現を抑制します。
- 協調的な制御:Sin3に結合することで、転写因子STICKYは自らが結合した配列近傍の遺伝子の活性化だけでなく、抑制/サイレンシングにも関与できると考えられます。
ドメインごとの重要性と機能的連動
| ドメイン | 予測される主な役割 | STICKY機能との関係 |
|---|---|---|
| bHLHドメイン | 標的配列へのDNA結合/ダイマー形成/発生・分化制御 | STICKYがゲノム内特定領域へ結合する“アドレス”機能 |
| Sin3相互作用ドメイン | Sin3複合体を介した遺伝子抑制/クロマチンリモデリング | STICKYが結合部位で抑制・サイレンシング作用を及ぼす |
理論的まとめ
- STICKYはbHLHドメインを介して特定のDNA配列(E-box)に結合し、遺伝子発現を直接制御する転写因子として作用すると予想される。
- Sin3相互作用ドメインの存在により、STICKYはSin3/HDAC複合体を標的遺伝子座にリクルートし、ヒストン脱アセチル化を通じてクロマチンを凝縮・転写抑制に導く「転写リプレッサー機能」を担うと考えられる。
- 両者のドメインが協調することで、STICKYは標的遺伝子の厳密な発現抑制や細胞分化のタイミング決定、あるいは環境応答など、多機能な調節因子として働く可能性が高い。
参考
以上より、STICKYは「特定配列結合―Sin3複合体リクルート型の転写抑制因子」として働く可能性が高く、両ドメインはいずれもその機能発揮に不可欠となるでしょう。
酵母ツーハイブリッドシステムは、既知のタンパク質やタンパク質ドメインに相互作用するタンパク質を同定するための有力な分子遺伝学的方法である。ここで、グルココルチコイド受容体(GR)が活性化ドメイン、DNA 結合ドメイン、リガンド結合能をもつ第二の活性化ドメインをもつモジュールタンパク質である証拠があったとしよう。さらに解析した結果,下垂体細胞では、このタンパク質はリガンドであるホルモンがない場合には細胞質にとどまっていることがわかったことから、これは他の阻害タンパク質と結合しているだろうと考察した。どのように酵母ツーハイブリッドシステムを用いて、GRが相互作用するタンパク質を同定することができるかを述べよ。また、GRにおいて、阻害タンパク質を特異的に結合するドメインを同定するにはどのようにすればよいか.
酵母ツーハイブリッドシステムを用いたGR相互作用タンパク質の同定法、および阻害タンパク質結合ドメインの解析法
酵母ツーハイブリッドシステム(Y2H)は、GR(グルココルチコイド受容体)のような多ドメインタンパク質が他のタンパク質と相互作用する相手や、特定ドメインを介した結合を明らかにするために有効に使われます。
GRが相互作用するタンパク質の同定方法
- ベイト(bait)構築
- 目的タンパク質(ここではGR全長または特定ドメイン)を、DNA結合ドメイン(DBD)をコードするベクターに融合させて発現させる(例:Gal4-DBD, LexA-DBD)。
- プレイ(prey)ライブラリの準備
- 相互作用パートナー候補となりうるcDNAライブラリ(例:下垂体由来など関心組織由来)を、転写活性化ドメイン(AD)をコードするベクターと融合して作成する。
- 酵母への導入・選抜
- ベイトとプレイの2種類のプラスミドを酵母細胞に共導入する(またはそれぞれ別の株に導入して交配させる)。
- 相互作用が生じた場合のみ、機能的な転写因子が再構成され、レポーター遺伝子(例:HIS3, LacZ)の発現によってスクリーニングされる。
- 陽性クローンの回収・シーケンス決定
GRの阻害タンパク質結合ドメインを特定する方法
- GRドメイン切断体(変異体)ベイトの作成
- 各ベイトごとのツーハイブリッドスクリーニング
- それぞれのGR切断体ベイトを用いて、同じcDNAライブラリまたは特定の阻害タンパク質候補のプレイと組み合わせてスクリーニングを行う。
- どのドメイン切断体で相互作用シグナル(レポーター発現)が得られるかによって、阻害タンパク質がGRのどの領域と結合しているかを絞り込む。
- ミニマルドメインの決定
- ドメイン間の境界付近でさらに小さい欠失変異体を作成し、細かくスクリーニングすることで、阻害タンパク質が結合するミニマル配列領域を明らかにできる。
まとめ
- 酵母ツーハイブリッド法は、GRが結合する未知のタンパク質(例えば細胞質保持型阻害タンパク質)を網羅的に探索・同定するのに有効な技術です。
- ドメイン別の欠失変異体を用いたツーハイブリッド解析により、GR内のどのドメインが阻害タンパク質と相互作用するかを特異的に同定できます。
- この手法を組み合わせることで、GRの細胞内局在制御や機能抑制メカニズムの分子基盤解明に役立ちます。
熱ショック遺伝子にコードされるタンパク質には、過酷な条件から他のタンパク質をすばやく守る働きをするものがある。このような遺伝子を制御するために進化した機構を述べよ。
熱ショック遺伝子は、細胞が熱などのストレスに晒された際に迅速に応答し、細胞を保護するタンパク質(熱ショックタンパク質、HSP)を合成します。この緊急事態に素早く対応するため、非常に巧妙な転写制御機構が進化してきました。
その最大の特徴は「転写の準備を済ませておき、いつでも発進できる状態で待機している」という点です。
熱ショック遺伝子の制御機構
この機構は、主に以下の2つの要素によって成り立っています。
- 待機状態のRNAポリメラーゼII(Paused RNA Polymerase II)
- ストレスがない平常時でも、熱ショック遺伝子のプロモーター領域にはRNAポリメラーゼIIが既に結合し、転写を始める準備ができています。
- しかし、ポリメラーゼは転写開始点から数十塩基進んだところで一旦停止(ポーズ)しており、それ以上進めないようにロックされています。
- これにより、転写開始の遅いステップである「転写装置の集合」をあらかじめ完了させておくことができます。
- 熱ショック転写因子(HSF1)の活性化
- 平常時、HSF1(Heat Shock Factor 1)は単量体として不活性な状態で細胞質に存在します。このとき、HSP90などの熱ショックタンパク質がHSF1に結合し、その活性を抑えています。
- 細胞が熱などのストレスを受けると、他のタンパク質が変性し始めます。すると、HSP90は変性タンパク質の修復に動員されるため、HSF1から離れます。
- HSF1は自由になると、三量体を形成して活性化し、核内へ移行します。
- 活性化したHSF1三量体は、熱ショック遺伝子のプロモーター上流にある熱ショックエレメント(HSE)という特定のDNA配列に結合します。
発現のスイッチオン
- HSF1がHSEに結合すると、それがシグナルとなり、停止していたRNAポリメラーゼIIのロックを解除する因子(例: P-TEFb)がリクルートされます。
- ロックが外れたRNAポリメラーゼIIは一斉に転写を再開し、HSPのmRNAを大量かつ迅速に合成します。
この機構の進化的意義
- 迅速性: 転写装置をゼロから組み立てる必要がなく、「発進許可」を出すだけなので、ストレスを感知してから数分という極めて短時間で応答できます。細胞の生存にとって、このスピードは決定的です。
- 効率性: 緊急時に必要な遺伝子群を協調的かつ強力に発現させることで、効率的に細胞を防御する体制を整えることができます。
| 状態 | RNAポリメラーゼIIの挙動 | HSF1の挙動 | 遺伝子発現 |
|---|---|---|---|
| 平常時 | プロモーターに結合し、停止(ポーズ)状態 | 不活性な単量体として細胞質に存在 | 抑制 |
| ストレス時 | 停止が解除され、転写を活発に再開 | 活性化して三量体を形成し、HSE配列に結合 | 誘導(活性化) |
このように、熱ショック遺伝子は「いつでも出動できるようエンジンをかけたまま待機する」という洗練された戦略をとることで、細胞の危機に即座に対応できるように進化してきました。
問題文引用元:東京化学同人 分子細胞生物学 第6版
コメント